Article | 26/11/2008
Faire parler l'ADN des fossiles
26/11/2008
Résumé
L'ADN des fossiles : des espoirs fous aux espoirs réels.
Table des matières
Ce texte a été commandé par le Conseil Général de l'Allier dans le cadre des travaux préparatoires au Centre Européen de Paléontologie qui va être construit à Gannat (03) dans les années qui viennent.
Ce texte est publié dans Planet-Terre avec l'accord du Conseil Général de l'Allier.
Faire revivre un fossile à partir de son ADN : un rêve de courte durée
Des dinosaures dans un parc d'attraction : à en juger par le succès reçu par le film Jurassic Park, l'effet serait garanti. Avant toute chose, il faudrait pour cela disposer de l'ADN de l'un de ces grands reptiles, c'est-à-dire de l'ADN tout droit sorti de l'ère secondaire ! Au bas mot, de l'ADN vieux de plus de soixante-cinq millions d'années ! Or, au milieu des années quatre-vingt, des scientifiques très sérieux avaient déjà fait sensation en faisant resurgir du passé des bribes de l'ADN d'un animal aujourd'hui disparu, le couagga, ainsi que d'une vieille momie égyptienne. Et si la promesse du film était possible ? Il y avait toutes les raisons d'y croire car les revues scientifiques les plus sérieuses alignaient très vite des unes toutes plus sensationnelles les unes que les autres. Partis des temps historiques, les scientifiques plongeaient très vite dans les temps préhistoriques (jusqu'à quelques dizaines de milliers d'années) et, semblait-il, l'ADN survivait même au-delà : jusqu'à une vingtaine de millions d'années semblaient indiquer les séquences obtenues à partir de feuilles fossilisées au fond d'un lac, ou les restes magnifiquement préservés d'insectes piégés dans l'ambre. Il n'y avait plus qu'une marche à gravir et l'on serait bientôt à l'ère des dinosaures ! Novembre 1994 : un tibia vieux de quatre-vingt millions d'années délivrait la première séquence d'ADN. Certes, avec ses quatre-vingt quatre nucléotides, on était bien loin de disposer des milliards de lettres nécessaires à l'écriture de son génome mais on pouvait somme toute commencer à rêver…
Figure 1. Rencontre avec des dinosaures... encore une fiction pour longtemps (Jurassic Park)
Image du film de Steven Spielberg, Jurassic Park, Universal Pictures
Néanmoins, le rêve allait être de courte durée puisque l'année suivante, en 1995, des études venaient nous remettre les pieds sur terre. On avait réussi à séquencer de l'ADN de dinosaures ou d'insectes préservés dans l'ambre, dites-vous ? Vraiment ? Car à y regarder de plus près, les séquences ressemblaient comme deux gouttes d'eau à des séquences de champignons et des séquences… humaines tout à fait actuelles ! Les résultats n'avaient donc rien de sensationnels : l'ADN obtenu n'était pas celui des fossiles mais plutôt celui d'organismes bien vivants. Ils avaient pour les premiers tout simplement élu domicile à la surface des vestiges ou pour les seconds, étudié le fossile d'un peu trop près et déposé leur propre ADN.
La fragilité de l'ADN et sa conservation
Le ciel se couvrait donc au-dessus du rêve jurassique et en réalité, il faudrait bientôt définitivement enterrer nos espoirs. Car, l'ADN est certes une molécule stable, mais elle n'est pas éternelle. Sa dégradation commence même aussitôt la mort d'un individu survenue ; les membranes de cellules cèdent en effet, les tissus se désorganisent et baignent dans leurs propres liquides. Bref, tout est réuni pour que des réactions d'hydrolyse se produisent. Résultat : les molécules d'ADN se fragmentent et certaines des lettres constitutives de l'information génétique sont peu à peu effacées. Ce n'est pas tout : les réactions d'oxydation poursuivent leurs irréversibles dégâts. Les chimistes estiment que dans ces conditions, une dizaine de milliers d'années peut suffire à venir à bout de la dernière molécule d'ADN. Ajoutez à cela les effets des organismes décomposeurs et vous comprendrez qu'il n'y a absolument aucune chance de retrouver de l'ADN à des époques aussi lointaines que celles des dinosaures. Jusqu'à quelle époque peut-on alors remonter ? Quelques dizaines de milliers d'années tout au plus. À moins que des conditions environnementales n'aient considérablement ralenti les réactions de dégradations, car alors peut-on atteindre à l'extrême limite quelques centaines de milliers d'années. De manière surprenante, de tels coffre-forts ne sont pas à rechercher du côté des vestiges conservés dans l'ambre dominicain ou des momies égyptiennes mais du côté des spécimens congelés dans les glaces de Sibérie ou du Groenland ou les pergélisols péri-arctiques. Car mieux que tout autre facteur, c'est le froid qui préserve le plus des outrages du temps.
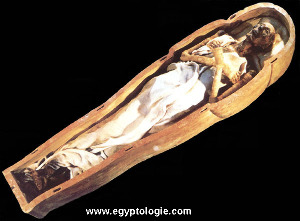
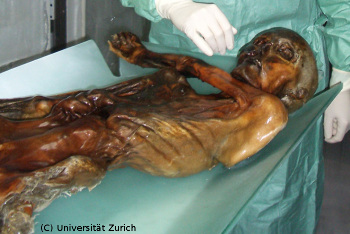
Source - © 2008 www.egyptologie.com (Rois / Ramsès II) Figure 2. Un exemple de corps momifié : Ramsès II | Source - © 2007 Universität Zurich Figure 3. Ötzi : conservé dans un glacier alpin Découvert en 1991 à plus de 3200 m d'altitude dans un glacier alpin. |
Source - © 2008 S.C. Schuster, Mammoth Genome Project, Pennsylvania State Univ. | Source - © 2006 L. Carion, www.carionmineraux.com Figure 5. Insectes pris dans de l'ambre |
Ainsi, les spécimens de Mammouth laineux - congelés entier dans les glaces - ou encore Ötzi - le chasseur retrouvé dans un glacier alpin plus de cinq mille ans après sa mort - contiennent-ils encore de l'ADN relativement bien conservé. Cela dit, ces conditions là sont plutôt exceptionnelles. Heureusement, bien d'autres vestiges peuvent encore receler des traces d'ADN, pourvu que leur environnement, par sa chaleur et son humidité, n'ait pas hâté le processus de dégradation. Des poils, des cheveux, des graines, des fragments de bois, mais surtout des os et des dents fossilisés ; voici le matériel de choix de l'apprenti paléogénéticien (chercheur d'ADN fossile). Et quelquefois, même, des vestiges plus saugrenus délivrent leurs précieuses informations. Tel est le cas des coprolithes (excréments fossilisés) qui ont par exemple permis de déterminer une partie des ingrédients qui avaient servi à préparer le repas d'un Américain mort deux mille cinq cents ans avant Jésus-Christ, ou encore trahi l'existence d'une espèce de paresseux méconnue et qui s'est éteinte lors du dernier épisode glaciaire, voilà douze mille ans.
Des conditions opératoires strictes, des techniques en évolution et de nouveaux horizons
Techniquement, récupérer de l'ADN à partir d'un reste fossile est une opération extrêmement délicate. Tout d'abord, ce qui reste de l'ADN de départ est mal-en-point. Notre ADN, et même celui qui est contenu dans la charcuterie de nos sandwichs, est donc infiniment en meilleur état. Il faut donc prendre les mesures les plus drastiques pour être sûrs de ne pas prendre le second (notre ADN) pour le premier (l'ADn des fossiles). Certes, l'erreur serait facile à repérer car la séquence de régions bien choisies de l'ADN peut servir, nous l'avons vu, à faire la différence entre les espèces. Mais qu'il serait dommage d'entreprendre une si contraignante et coûteuse analyse pour finir par mettre les résultats à la poubelle ! Concrètement, « mesures drastiques » signifient travailler à la fois avec un équipement stérile (blouse, charlottes, gants, surchausses à usage unique) et dans des locaux aussi exempts d'ADN que possible. Pour cela, les surfaces sont nettoyées quotidiennement à l'eau de javel et en permanence exposées aux rayons ultraviolets. De plus, l'atmosphère est maintenue en surpression, afin d'éviter toute entrée indésirable d'ADN depuis l'extérieur. Toutes ces précautions visent à minimiser la part des contaminations par l'ADN actuel.
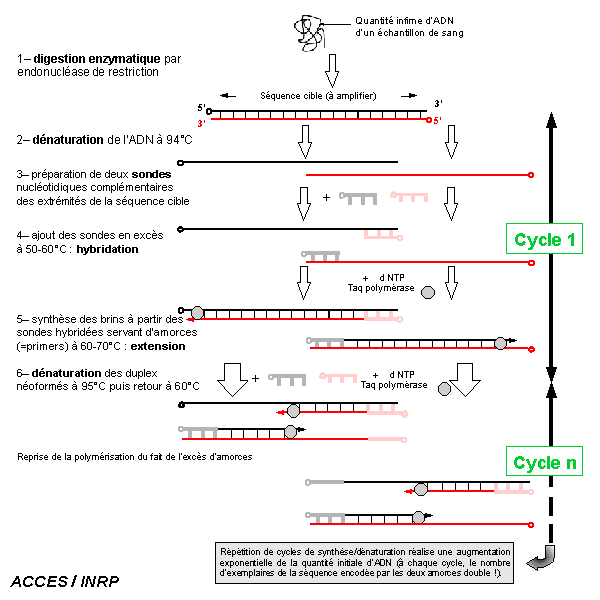
Source - © 2008 ACCES / INRP
Figure 6. Schéma de principe de la technique de PCR
PCR : Polymerase Chain Reaction (amplification en chaîne par polymérase). Cette technique permet d'obtenir, à partir d'un échantillon complexe et peu abondant, d'importantes quantités d'un fragment d'ADN (environ 1 million de copies en quelques heures). C'est, généralement suffisant pour une utilisation ultérieure.
Voir aussi : Présentation de la PCR (ENS de Lyon).
Voilà pour le matériel. Voyons maintenant le protocole opératoire. Tout commence par le broyage d'un échantillon d'environ cinq cent milligrammes d'un fossile. Ensuite, la poudre est mise à incuber plusieurs heures dans un tampon facilitant la dégradation des matériaux indésirables (protéines par exemple), et s'il s'agit de la poudre d'os ou de dents, d'un agent dissociant la matrice calcifiée. Résultat : l'ADN, s'il est encore préservé, est libéré dans le tampon. La suite des opérations ne vise donc plus qu'à le précipiter et concentrer dans un volume facilitant sa manipulation. Cependant, à ce stade, ne subsistent que de rares traces d'ADN en mauvais état, soit autant de mauvais substrat relativement réfractaires aux techniques classiques de séquençage. Il faut donc généralement viser une région précise du génome et la photocopier des milliards de fois avant d'espérer déterminer la séquence souhaitée. C'est précisément l'objet de la PCR (Polymerase Chain Reaction, ou Réaction de Polymérisation en Chaîne), une technique de biologie moléculaire mise au point au milieu des années 1980, et désormais courante dans les laboratoires. Cependant, si la technique présente l'avantage de ramener en quantités suffisantes des substrats qui ne subsistaient plus qu'à l'état de traces, la technique peut, si l'on n'y prend garde, tout aussi bien multiplier ad infinitum nos indésirables contaminants. L'étape est donc particulièrement risquée et délicate et nécessite de nombreux contrôles de qualité. Mais on n'est pas encore au bout de ses peines : les molécules d'ADN fossile étant particulièrement abîmées, les photocopier engendre de très nombreuses coquilles. Il faudra donc encore dépouiller les exemplaires photocopiés de ces coquilles avant de recomposer la séquence finale. Et gage ultime d'authenticité, la séquence doit être reproduite plusieurs fois, et de préférence, dans un laboratoire indépendant.
Source - © 2008 S.C. Schuster, Pennsylvania State Univ.
Long et périlleux est donc le chemin qui mène à une séquence d'ADN fossile. Et pourtant, combien cette dernière est courte ; en effet, dans les fossiles, l'ADN n'est pas conservé sous la forme de longs chromosomes intègres mais sous la forme de très courts fragments. Aussi ne peut-on généralement espérer obtenir suite à une photocopie qu'un fragment long de cent cinquante à deux cents nucléotides ! Inutile de préciser que dans ces conditions, plusieurs fragments seront nécessaires avant d'obtenir une quantité d'information suffisante pour répondre aux questions posées. Autre point très important : le plus souvent, les gènes ciblés ne correspondent pas à des gènes portés par les chromosomes. Ils sont hébergés par un autre type d'ADN qui dans nos cellules, n'est pas contenu dans le noyau mais dans d'autres compartiments : les mitochondries (il s'agit donc de l'ADN mitochondrial). C'est au sein des mitochondries que s'opère les réactions de respiration dans nos cellules. Certes, cet ADN-là est-il généralement court - environ seize mille nucléotides chez l'Homme – mais il présente plusieurs avantages : d'une part, son évolution est rapide si bien qu'il sera possible de suivre des événements du passé avec une bonne résolution et d'autre part, chaque cellule présente au bas mot un millier de mitochondries. Autant dire, un millier de molécules d'ADN mitochondrial. Or, chaque chromosome dans notre noyau n'est présent qu'en deux exemplaires. Ainsi, en ciblant spécifiquement l'ADN mitochondrial, les paléogénéticiens multiplient-ils les chances de réussir à mettre la main sur un bout d'information encore conservée. L'ADN mitochondrial présente toutefois une particularité notable : il ne se transmet que de mère à enfant. Ainsi, il sera d'une totale inutilité pour effectuer d'éventuels tests de paternité.
Mi-2006, une modification subtile de la technique de PCR a cependant permis de s'affranchir de l'ADN mitochondrial en permettant à des paléogénéticiens de reconstituer la séquence totale d'un des gènes responsables de la couleur des poils du Mammouth laineux. Or, fin décembre 2005, une nouvelle technique de séquençage avait déjà révélé tout son potentiel à la communauté des paléogénéticiens. Grâce à elle, il a été possible d'obtenir plus de treize millions de nucléotides de gènes localisés sur les chromosomes du Mammouth laineux ! Ainsi, les gènes portés par nos chromosomes devraient-ils à l'avenir être de plus en plus la cible des paléogénéticiens. Avec eux s'ouvriront toute une variété de questions nouvelles, par exemple relatives à l'allure des espèces préhistoriques, comme leur couleur de peau par exemple. Aussi, certains laboratoires très sérieux, comme celui dirigé par Svante Pääbo à Leipzig, annoncent-ils déjà le séquençage du génome complet de l'Homme de Néandertal ! Celui du Mammouth est quant à lui déjà en cours. Autant de promesses qui rendent l'actualité de la paléogénétique particulièrement attrayante. Mais d'ores et déjà, cette discipline a permis de résoudre des énigmes très intéressantes. Voyons-en quelques unes.
Source - © 2007 NTV, Russian Television Channel (voir Futura Siences)
Figure 8. Un jeune mammouth congelé extrait du pergélisol, Sibérie
Petite femelle de 6 mois, pesant 65 kg, haute de 85 cm, longue de 130 cm. Gelée de puis 10 000 ans.
Taxonomie des moas
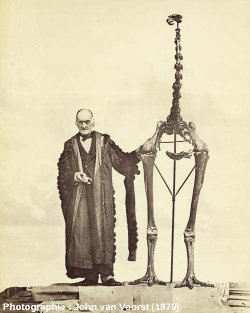
Richard Owen est un paléontologue anglais du XIXème siècle, connu pour avoir inventé le terme "dinosaure". Au cours de sa carrière, il s'est aussi intéressé à la classification d'oiseaux fossiles : les moas. Ces derniers marchaient, courraient mais ne savaient pas voler. Ils peuplaient la Nouvelle-Zélande il y a encore peu de temps mais la venue de l'Homme et de trop nombreux épisodes de surchasses drastiques leur ont été fatal. Selon Owen, les nombreux vestiges fossiles de Moas pouvaient être classés selon leur taille. Ainsi, il pouvait définir différentes espèces, allant de celles présentant des fémurs courts à celles présentant les fémurs les plus longs.
Figure 9. Richard Owen posant à côté du plus grand squelette de moa, Dinornis giganteus (robustus)
R. Owen tient ici à la main, le premier fragment étudié qu'il attribua 40 ans plus tôt à un tel oiseau.
Plus de cent cinquante ans plus tard, des paléogénéticiens d'Oxford valideraient-ils avec l'ADN ce système de classification ? Si Owen disait vrai, on retrouverait trois espèces parmi les moas Dinornis. Quelques dizaines d'échantillons et séquences d'ADN mitochondrial plus tard, les trois groupes se faisaient toujours désirer. Tout au plus, l'ADN mitochondrial arrivait-il à distinguer deux groupes, correspondant aux populations de l'île du Nord et de l'île du Sud, séparées par le détroit de Cook. Bref, une seule espèce, et non les trois escomptées. À quoi pouvait donc correspondre les différences de taille entre spécimens si elles n'indiquaient pas une différence d'espèces ? À des différences entre sexes à n'en pas douter puisque les fémurs longs montraient toujours des fragments de gènes du chromosome Z et du chromosome W (chez les oiseaux, les femelles sont ZW et les mâles ZZ contrairement à notre espèce où ce sont les mâles qui sont XY et les femelles XX). Quant aux fémurs courts, le diagnostic ZZ assurait qu'il s'agissait de mâles. Owen avait donc fait des deux sexes d'une même espèce deux espèces distinctes (la détermination du sexe est délicate chez les oiseaux, même celles qui vivent aujourd'hui) ! Et à dire vrai, les grands mâles et les petites femelles constituaient-ils pour lui une troisième. Loin d'être anecdotique, cette étude révèle l'une des difficultés fondamentales de la paléontologie : définir des regroupements de vestiges qui correspondent à des espèces vraies, et non à des ressemblances fortuites, et concomitamment ne pas multiplier les noms d'espèces en s'appuyant sur des différences non significatives (comme les différences entre sexes). Il faut dire qu'une espèce se définit généralement comme l'ensemble des individus capables de se reproduire et d'avoir des descendants eux-mêmes fertiles et qu'un paléontologue peut difficilement faire se reproduire des os ! L'ADN peut désormais apporter une solution au problème !
Nos liens avec l'Homme de Néandertal
Que n'a-t-on pas dit sur l'Homme de Néandertal ? « Bête sauvage (..) à peine sorti de l'animalité », il en a fallu du temps pour voir sa réhabilitation. Car, depuis sa reconnaissance comme un Homme fossile au milieu du XIXème siècle, ses vestiges n'ont cessé de nous renvoyer à notre propre humanité. Aujourd'hui, on reconnaît volontiers son intelligence (son cerveau était d'ailleurs plus gros que le nôtre), sa croyance en un "au-delà" (il enterrait ses morts, quelquefois même avec des offrandes) et son habileté technique (il disposait d'une industrie lithique relativement sophistiquée) car ce lointain cousin est connu par plus de trois cent spécimens. Mais au fait, s'agit-il vraiment d'un lointain cousin ? N'était-il pas plutôt notre frère ? Il faut dire que lorsque l'Homme moderne découvre l'Europe – il y a environ quarante mille ans de cela – l'Homme de Néandertal y est déjà établi depuis au moins cent cinquante mille ans. L'Homme de Néandertal ne disparaîtra que dix mille ans plus tard, ce qui a donc laissé une longue période de cohabitation où l'un et l'autre ont pu se croiser. Dans ce cas, nous aurions, nous Hommes européens modernes, un peu de sang néandertalien (des gènes en réalité) en nous. Certains fossiles, comme l'enfant de Lagar Velho (Portugal), datant de vingt-cinq mille ans semblent l'indiquer car ils témoignent d'une mosaïque de caractères typiquement néandertaliens ou modernes. Mais d'autres indices semblent au contraire indiquer une extinction sans mélange. À la fin des années 1990, la paléogénétique exhumait une première séquence de l'ADN mitochondrial. Elle provenait du spécimen type de Feldhofer découvert en 1856 dans la vallée de la Neander, non loin de Düsseldorf. Dix ans plus tard, une dizaine d'autres spécimens ont délivré de nouvelles séquences. Toutes pointent vers la même conclusion : de telles séquences ne sont connues chez aucun de nos contemporains. Difficile dans ces conditions d'imaginer un quelconque croisement. Cependant, difficile pour l'instant de l'exclure définitivement sur la base de si peu de données : des modèles bioinformatiques savants estiment cependant que, si elle a eu lieu, elle resta absolument modeste. Gageons que l'analyse paléogénétique de nouveaux spécimens et le génome néandertalien annoncé sauront faire avancer la question. D'ailleurs, la séquence d'un spécimen daté de cent mille ans provenant de la grotte de Scladina n'a-t-elle pas déjà révélé une séquence légèrement différente des autres. Affaire à suivre !
Source - © 2006 Oxford University Press, dans Global Change (Univ. of Michigan)
Pour aller plus loin ...
Ludovic ORLANDO, 2005. L'anti-Jurassic Park. Faire parler l'ADN fossile. Éditions Belin/Pour la Science, collections Regards sur la Science, 272pp.