Article | 23/09/2003
Les traces d'ADN fossile
23/09/2003
Résumé
Les processus de dégradation de l'ADN et les plus vieilles traces connues d'ADN fossile.
Table des matières
Question
Objet : Fossiles et ADN Date : Mar, 24 Apr 2003 09:42:20 De : J. F.
« Quel est l'âge du plus ancien animal, sur lequel un séquençage d'ADN a pu être réalisé, et sur quel matériel (os, phanère,...) ? Même question pour les végétaux ? »
Réponse
L'ADN se conserve mal ! Il est normalement dégradé au cours de la décomposition de l'organisme par des enzymes endogènes (les nucléases), elle-mêmes libérées par la destruction des cellules.
À propos de la conservation de la molécule d'ADN : une durée de vie théorique de 100.000 ans
Voici ce qu'écrivent à ce sujet Hofreiter et al. dans leur article Ancient DNA publié dans Nature - Reviews Genetics, Vol. 2, n° 5, pp. 353-359, 2001 (doi : 10.1038/35072071) :
En supposant des circonstances favorables (dessiccation rapide, basses températures ou fortes concentrations en sels), les nucléases peuvent être elle-mêmes détruites avant que tout l'ADN n'ait été endommagé ou digéré et réduit à ses mononucléotides.
Dans ce cas, des processus de dégradation plus lents peuvent affecter alors l'ADN.
Source - © 2001 Nature Reviews in Genetics
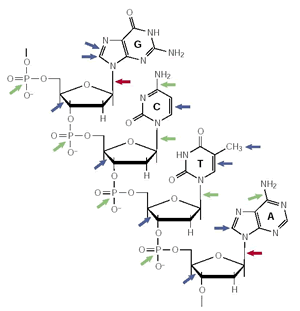
Figure 1. Dommages affectant une molécule d'ADN fossile
Un court segment d'un seul brin de la double hélice d'ADN est représenté avec les quatre bases azotées classiques. Les principaux emplacements des dommages (dépurination) sont indiqués par les flèches rouges. Des emplacements qui peuvent subir une attaque hydrolytique sont indiqués par les flèches vertes et ceux pouvant subir une oxydation sont indiquées par les flèches bleues. G : guanine ; C : cytosine ; T : thymine ; A : adénine.
Par exemple, l'oxydation (flèches bleues) ou les effets directs et indirects des radiations de fond vont modifier les bases azotées et le squelette de phosphate et de sucres de l'ADN. De plus, désamination, dépurination (flèches rouges) et autres processus hydrolytiques (flèches vertes) vont déstabiliser et briser les molécules d'ADN.
Tous ces phénomènes compliquent la récupération des séquences anciennes d'ADN. Par exemple, une forte proportion des résidus cytosine (C) et thymine (T) des extraits tissulaires anciens est modifiée par oxydation (flèches bleues) en hydantoïnes, qui bloquent les ADN polymérases et donc la PCR : le séquençage de l'ADN devient donc impossible.
En supposant des concentrations physiologiques en sels, un pH neutre et une température de 15°C, il faut environ 100 000 ans aux attaques hydrolytiques pour détruire tout l'ADN que l'on pourrait espérer récupérer. Certaines conditions environnementales, telles que de basses températures, peuvent étendre cette durée-limite, alors que d'autres la réduisent.
Source - © 2001 Hofreiter M. et al., Nature Reviews in Genetics
Figure 2. Coprolite âgé de 20.000 ans
À partir de cet échantillon de coprolite âgé de 20.000 ans, des séquences d'ADN ont été déterminées pour l'animal mais aussi pour les espèces vivantes qui l'ont alimenté.
À propos de découvertes d'ADN fossile de plus de 50.000 ans
Le plus vieil ADN reconnu comme authentique provient de l'os d'un mammouth congelé dans le permafrost de Sibérie, dont l'âge est estimé à environ 50 000 ans.
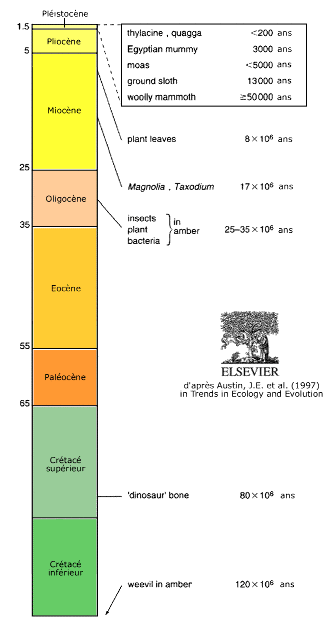
Les séquences d'ADN de plus d'un million d'années sont qualifiées « d'antédiluviennes ». Toutes les séquences de ce type soit n'ont pas pu être reproduites, soit provenaient d'une contamination par une source externe, identifiée par la suite.
Source - © 1997 J.E. Austin et al., Trends in Ecology and Evolution
On constate que la majorité des découvertes se situe dans les 50 000 dernières années, ce qui est en accord avec la durée de vie théorique de la molécule d'ADN.
Ainsi, les séquences d'ADN issues d'inclusions dans l'ambre datant du Miocène (de 5 à 24 millions d'années) ne peuvent pas être obtenues. On a pu montrer que ces inclusions sont fortement modifiées par les processus diagénétiques. De même, on n'a pas pu reproduire les séquences provenant de fossiles compressés de plantes Miocène.
En Ardèche, du matériel fossile a été préservé dans un dépôt de diatomite de 8 à 8,5 millions d'années. Des séquences d'ADN chloroplastique ont été retrouvées dans 11 feuilles fossiles bien conservées. La silice, qui compose la diatomite, est susceptible de se lier à l'ADN et ainsi de le protéger de la dégradation. Cependant, on n'obtient pas de bonne corrélation entre l'identité des plantes fossiles et les séquences d'ADN obtenues.
Sur le site de Clarkia (Nord de l'Idaho, USA), de l'ADN a été récupéré par deux groupes indépendants à partir de feuilles bien préservées de Magnolia et de Taxodium âgées de 17 à 20 millions d'années. L'état de préservation des biopolymères (comme les polysaccharides et les protéines) de ces feuilles n'a pas permis la reconstitution de l'ADN initial.
D'autres chercheurs ont conduit des travaux de ce type, mais aucun ADN de plante n'a pu être récupéré.
Quant aux séquences extraites d'un os de dinosaure du Crétacé, elles se sont révélées dérivées d'une segment d'ADN mitochondrial inséré dans le génôme humain (un pseudogène).