Article | 13/11/2025
De l'ADN au fossile. Les dénisoviens, quand les paléomolécules guident la paléontologie
13/11/2025
Auteur(s) / Autrice(s) :
Publié par :
- Olivier DequinceyENS de Lyon / DGESCO
Résumé
De l’analyse morpho-anatomique à l’analyse du génome voire du protéome pour décrire des fossiles et les attribuer à une espèce d’hominine.
Résumé étendu. Il y a quinze ans, en avril 2010, la petite communauté de la paléo-anthropologie était secouée par un séisme inattendu : la paléogénétique en plein développement dévoilait dans la revue Nature un résultat spectaculaire, la découverte, sur la base d'un génome mitochondrial, d'une forme humaine inconnue dans le registre fossile et pourtant contemporaine des néandertaliens et d'Homo sapiens. Quinze ans plus tard, les données moléculaires se sont accumulées et ont éclairé la répartition géographique de ces hominines, dénommés “dénisoviens” mais toujours dépourvus de nom d'espèce, leur démographie et, quoiqu'avec beaucoup d'incertitude, quelques éléments de leur anatomie. Ça n'est que dans ces dernières années que des fossiles ont enfin pu leur être formellement attribués : après des résultats similaires obtenus sur des mâchoires isolées, une équipe chinoise vient d'identifier des protéines et de l'ADN mitochondrial dénisoviens dans un crâne fossile, donnant ainsi un visage à cette population énigmatique. Cet article tente de balayer les principaux résultats de quinze ans de recherche sur cet « hominine invisible », qui illustre aussi l'importance prise par les méthodes de récupération et d'analyse des molécules anciennes, acides nucléiques et protéines.
Introduction
Depuis le début du XXIe siècle, la paléo-anthropologie a connu plusieurs bouleversements, qui l'ont remise sur le devant de la scène scientifique et médiatique. Ses avancées figurent à nouveau en bonne place dans les revues de vulgarisation scientifique et font aussi l'objet d'articles et de chroniques dans les rubriques scientifiques des journaux et des médias audiovisuels, comme c'était le cas un siècle auparavant, quand ceux que l'on nommait encore seulement anthropologues et paléontologues cherchaient le « chainon manquant » entre l'humain et les grands singes en Afrique (suivant l'hypothèse de Charles Darwin) et en Asie du Sud-Est (sous l'influence des théories d'Ernst Haeckel). De même que le « chainon manquant » devenait un personnage de feuilleton journalistique, de nouvelles ou de romans pour Maurice Leblanc ou Gaston Leroux[1], néandertaliens et Homo erectus sont désormais des personnages de romans policiers et de science-fiction[2]. Ces bouleversements ont commencé en 2000 avec la découverte des fossiles qui repoussaient l'origine de la « branche humaine » au-delà de 4 millions d'années (Ma), à environ 7 Ma ; ils se sont poursuivi avec la description de nouveaux australopithèques (Australopithecus sediba, 2015) et de nouveaux représentants du genre Homo (Homo georgicus, 2002) ; la mise au jour de fossiles étranges combinant des caractères archaïques et des âges étonnamment récents (Homo floresiensis en Asie du Sud-Est en 2004, puis Homo luzonensis en 2019, Homo naledi en Afrique du Sud en 2015). Mais c'est aussi le développement rapide de la paléogénétique qui a transformé ce domaine de recherche, forçant à la coopération paléontologues de terrain, généticiens en salle blanche et nouveaux spécialistes de bio-informatique. La découverte puis l'étude d'une nouvelle branche du buisson humain, fondée quasi-exclusivement sur l'analyse de molécules extraites de restes fossiles très peu spectaculaires, est devenu l'exemple emblématique de ce bouleversement.
Source - © 2021 — Ji et al. [ 24 ]
Deux analyses, protéomique et génétique, publiées en juin 2025 le rattachent aux dénisoviens (Fu et al. 2025a[12], 2025b[13]), confirmant les soupçons de certains paléoanthropologues (Hublin, 2021[20]).
Cet article vise à brosser un rapide bilan de la floraison de résultats obtenus en quinze années sur cette nouvelle population d'hominines, les dénisoviens, toujours dépourvus de nom d'espèce officiel – faute d'un fossile holotype – et alors que viennent de paraitre deux articles qui attestent, sur la base d'analyses paléogénétiques et paléoprotéomiques, de l'appartenance à ce groupe d'un fossile spectaculaire, un crâne très bien conservé.
L'ADN dénisovien, une affirmation spectaculaire de l'apport scientifique de la paléogénétique
Des bribes d'ADN mitochondrial ancien aux génomes complets
Les premières analyses d'ADN ancien, extrait de restes archéologiques ou fossiles, datent maintenant d'une quarantaine d'années, avec le publication le 15 novembre 1984, dans la revue Nature (Higuchi et al., 1984 [18]), d'une séquence d'ADN mitochondrial (ADNmt) de quagga, un équidé dont le dernier représentant s'est éteint en 1883. Mais cet ADNmt n'était qu'une fraction de matériel génétique dégradé, laborieusement obtenu en clonant l'ADN récupéré sur le spécimen dans des cultures bactériennes. La mise au point de la PCR (Polymerase Chain Reaction) dans la même décennie améliora largement la sensibilité de la méthode (c'est-à-dire sa capacité à récupérer et à multiplier une quantité d'ADNmt bien plus petite qu'auparavant), mais s'accompagna aussi d'un risque accru de contamination par l'ADN moderne ambiant. Vingt ans plus tard, une revue des progrès accomplis dans ce domaine soulignait encore combien ces analyses restaient compliquées à produire et sujettes à de fréquentes remises en question, en raison de contaminations du matériel ancien par de l'ADN récent.
The field is still regularly marred by erroneous reports, which underestimate the extent of contamination within laboratories and samples themselves. Traduction proposée. « Ce champ [de recherche] est encore régulièrement entaché par des rapports erronés, qui sous-estiment l'étendue de la contamination dans les laboratoires et dans les échantillons eux-mêmes. » | ||
| --Willerslev et Cooper, 2004 [48] | ||
Néanmoins, ces difficultés ont aussi contribué à mieux comprendre les processus de dégradation post-mortem des molécules biologiques et à développer des protocoles pour les prendre en compte. Dans une autre revue parue la même année, le futur prix Nobel et pionnier de ces études Svante Pääbo estimait encore prudemment que :
the study of ancient DNA sequences has had relatively limited impact on our understanding of recent human history, and this situation is unlikely to change in the near future. Traduction proposée. « L'étude des séquences d'ADN ancien n'a eu qu'une influence assez limitée sur notre compréhension de l'histoire humaine récente, et il est peu probable que cette situation change dans un futur proche. » | ||
| --Pääbo et al., 2004 [37] | ||
Pourtant, les recherches et les analyses d'ADN ancien se sont développées rapidement et ont évidemment abordé l'histoire humaine. Mais elles se sont d'abord concentrées sur l'ADN mitrochondrial, plus abondant et plus facile à séquencer. Cette approche a été notamment mobilisée pour trancher le vieux débat du statut taxinomique des néandertaliens vis-à-vis des Homo sapiens. La paléogénétique promettait d'apporter des données quantifiées sur l'éloignement génétique des deux populations, donc sur le statut d'espèce ou de sous-espèce des néandertaliens : Homo neanderthalensis ou Homo sapiens neanderthalensis ? La comparaison d'ADN mitochondrial récupéré sur des néandertaliens, des Homo sapiens “archaïques” datés d'une vingtaine de milliers d'années et des humains actuels montraient une nette distinction entre les séquences des premiers et ceux des deux autres (Caramelli et al., 2003 [6]), ce qui semblait signifier une séparation ancienne entre néandertaliens et sapiens et une très faible potentialité d'hybridation entre les deux groupes, qui pouvait justifier d'en faire deux espèces distinctes.
Le séquençage des génomes mitochondriaux puis nucléaires s'est poursuivi dans la première décennie du siècle, mais restait fastidieux et couteux, malgré le développement d'un protocole fiable et efficace, la méthode Sanger. Cette technique consistait à amplifier par PCR l'ADN étudié, à le découper en fragments de tailles variées par des enzymes (endonucléases), à marquer ces fragments par des groupements fluorescents, puis à faire synthétiser de nouveaux fragments par des ADN polymérases en leur fournissant, en plus des nucléotides normaux, un nucléotide débarrassé de deux groupements -OH (un didésoxynucléotide : ddA, ddG, ddT ou ddC). L'incorporation de ce composant artificiel dans le nouveau brin stoppe sa synthèse. Les morceaux d'ADN de différentes tailles et portant l'un ou l'autre des didésoxynucléotides étaient ensuite triés et détectés par chromatographie ; pour plus de détails, voir, sur Planet-Vie, l'article Le séquençage des génomes de G. Furelaud (2004[15]).
Des techniques plus rapides et plus automatisées ont vu le jour dans cette même décennie 2000-2010 ; plusieurs technologies concurrentes se sont alors disputé ce marché nouveau et prometteur. L'année 2010 a vu ces technologies arriver à maturité, réduisant drastiquement à la fois la durée et le cout d'un séquençage. Comme le décrit l'article de Van Dijk et Thermes, 2021 [47] sur La révolution de la génomique, l'une de ces options, la technologie Illumina, l'a emporté sur les autres et est aujourd'hui la plus largement employée dans les laboratoires. Ces nouveautés ont permis non seulement de séquencer beaucoup plus vite, mais aussi d'obtenir les séquences de molécules d'ADN nucléaires complètes, donnant accès à l'intégralité du génome nucléaire, d'où l'installation du néologisme « génomique » pour qualifier cette nouvelle voie de recherche. De même que s'impose désormais les mots « protéome » et « protéomique » pour désigner respectivement l'intégralité des protéines d'un organisme et son étude, ou celui de « transcriptome » (l'ensemble des ARN transcrits à partir de l'ADN dans une cellule, un tissu ou un organisme entier). Toutes ces collections de molécules (génome, protéome, transcriptome) et les disciplines qui s'en emparent ont pris leur essor grâce aux avancées techniques dans le séquençage et l'analyse de ces molécules d'une part, et à l'émergence des capacités de stockage et de traitement de ces énormes jeux de données, d'autre part. Et bien sûr à l'effondrement du cout de ces opérations.
L'analyse des génomes actuels a été presque aussitôt complétée par celle des génomes anciens : le premier génome mitochondrial ancien complet a été publié en 2001 (Orlando et al., 2021[36]). Mais c'est là aussi vers 2010 qu'ont commencé à se multiplier les séquençages d'ADN ancien, mitochondriaux puis nucléaires, jusqu'aux génomes nucléaires complets d'organismes fossiles : comme l'indique la figure 2, les publications de paléogénomes entiers (d'humains) ont augmenté considérablement, accélérant à partir de 2014 puis plus encore depuis 2017, où leur nombre a été multiplié par plus de sept en seulement trois ans. De plus, outre leur vitesse démultipliée, les séquenceurs automatiques ont très nettement accru la sensibilité des analyses : capables de retenir et de multiplier le moindre fragment d'ADN encore présent dans un échantillon, ils ont permis de diminuer la quantité minimum de matériel osseux ou dentaire nécessaire à l'obtention d'une séquence. Des structures fossiles de taille réduite ou peu informatives par leur morpho-anatomie deviennent donc des sources potentielles de matériel génétique. D'autant plus que chaque position du génome récupéré est désormais « lu » plusieurs fois, augmentant ainsi la fiabilité du résultat.
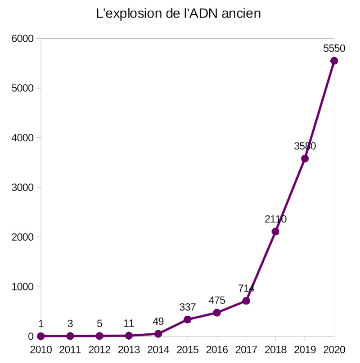
Source - © - — David Reich Lab
Le graphe montre le nombre cumulé de génomes d'individus anciens publiés.
La découverte (génétique) des dénisoviens
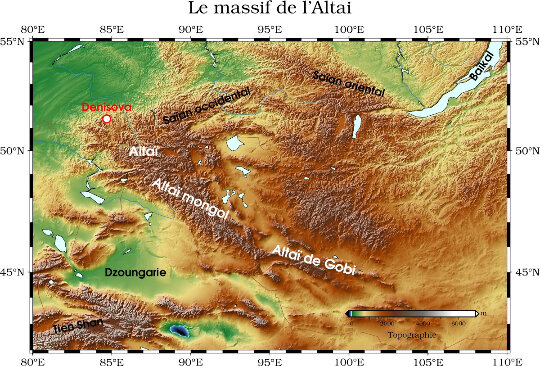
C'est néanmoins une analyse d'ADN mitochondrial ancien qui, la première, a dévoilé l'existence d'une population d'hominine jusque là inconnue. À cette date, on ne disposait pour comparaison que de 10 000 génomes d'humains modernes et de 7 génomes (mitochondriaux) néandertaliens. Cet ADN provenait d'une phalange distale retrouvée en 2008 dans les sédiments d'une grotte de l'Altaï, en Sibérie, dite « grotte de Denisova » (84°40'E et 51°23'N), du nom d'un ermite, Denis, qui y aurait trouvé refuge au XVIIIe siècle (localisation figures 5). L'examen de l'os permettait seulement de l'attribuer à un ou une enfant (figure 3). Cette grotte et ses alentours avaient déjà livré des ossements d'humain moderne (Homo sapiens) et de néandertaliens (Homo neanderthalensis) (voir plus loin). C'est d'ailleurs du matériel d'une autre grotte altaïque, celle d'Okladnikov (84°20'E et 51°40'N), que des chercheurs de l'institut Max Planck d'anthropologie évolutionnaire de Leipzig et de l'institut de génomique fonctionnelle (IGFL) de Lyon avaient précédemment extrait de l'ADN mitochondrial néandertalien, démontrant ainsi que l'aire de répartition d'Homo neanderthalensis s'étendait bien plus loin à l'Est que ce que l'on supposait jusqu'alors (Krause et al., 2007[26]). C'est à l'institut Max Planck que fut envoyée la petite phalange de Denisova. Et si l'extraction de l'ADNmt néandertalien d'Okladnikov publiée en 2007 avait nécessité 200 mg de poudre d'os pour un séquençage réalisé avec les protocoles classiques, celle de l'ADNmt de Denisova, issue d'un séquenceur de nouvelle génération Illumina et publiée en avril 2010, n'a demandé que 30 mg de matériau (Krause et al., 2010[25]). La comparaison de cet ADNmt à ceux d'humains actuels, d'Homo sapiens fossiles et de néandertaliens dévoila des différences inattendues, qui distinguait cet individu des sapiens comme des néandertaliens. La phylogénie obtenue à partir de ces séquences plaçait l'ADNmt de l'enfant de Denisova sur une branche isolée, séparée de celui des autres humains d'Eurasie depuis 1 million d'années, bien avant l'ancêtre commun aux néandertaliens et aux sapiens (donc aussi éloigné – évolutivement parlant – des premiers que des seconds, figure 4).
 Source - © 2010 — Reich et al. [ 41 ] , Max Planck Institute for Evolutionary Anthropology |
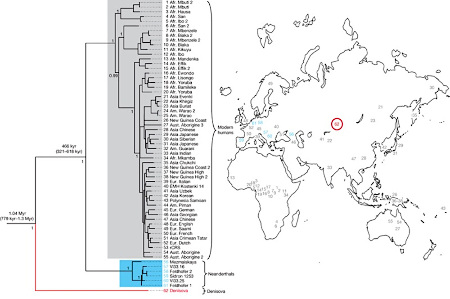 Source - © 2010 — Krause et al. [ 25 ] L'individu de Denisova se place en groupe-frère de l'ensemble (clade) regroupant néandertaliens (en bleu) et sapiens (en gris). Le cercle rouge sur la carte repère la grotte de Denisova. |
Les dénisoviens dévoilés à travers la paléogénétique
Les dénisoviens, cousins d'Asie des néandertaliens
Huit mois après la publication de l'ADN mitochondrial dénisovien, le laboratoire de Leipzig publiait l'analyse d'une nouvelle séquence d'ADN, toujours extraite de la même phalange mais il s'agissait cette fois-ci du génome nucléaire de cette fillette désormais cataloguée Denisova 3 (Reich et al., 2010[41]). La quantité d'ADN original récupérée dans ce tout petit os de 40 mg s'est en effet avérée étonnamment élevée (même si la quasi-totalité du fossile a disparu dans l'opération), et surtout très bien conservée, sans doute en raison des températures constamment basses qui règnent dans la grotte. Les auteurs de l'étude ont estimé ainsi que l'ADN de l'individu représentait 70 % de l'ADN total obtenu, les 30 % restant correspondant à de l'ADN microbien (alors que ces proportions sont généralement inversées, et souvent encore plus défavorables à l'ADN non-microbien). En conséquence, la couverture de ce séquençage était de presque 2 fois (1,9 fois)[3], ce qui revient à dire que chaque position de la séquence a été retrouvée, en moyenne, 1,9 fois, ce qui parait peu comparé aux résultats obtenus avec un génome actuel, mais remarquable pour un matériel dont l'âge dépasserait les 55 000 ans (Jacobs et al., 2019[22]).
Ce nouvel article revenait d'abord sur les conclusions de l'étude précédente, à la lumière des données de l'ADN nucléaire.
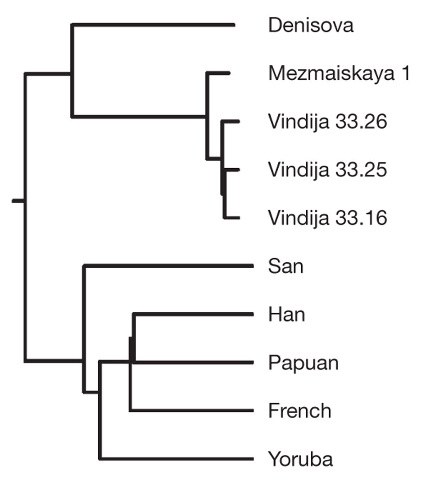
En comparant les régions de cet ADN au génome humain de référence[4], et en se limitant aux portions du génome humain d'ascendance africaine, c'est-à-dire aux régions qui ne comportent pas de gènes néandertaliens issus d'une hybridation sapiens - néandertalien en Eurasie, l'équipe a estimé le degré de divergence de l'ADN dénisovien par rapport à celui de l'humain actuel. Elle en a conclu que cette divergence est d'ampleur comparable à celle mesurée entre les génomes néandertalien et sapiens, alors que l'ADN mitochondrial suggérait une différence beaucoup plus importante ; ce premier résultat plaidait pour un rapprochement entre dénisoviens et néandertaliens. En alignant ensuite l'ADN dénisovien sur toutes les séquences néandertaliennes disponibles à l'époque (c'est-à-dire trois individus de Croatie et trois autres de Russie, d'Allemagne et d'Espagne) et sur des génomes humains de référence de différentes provenances géographiques (deux africains, un San et un Yoruba, un chinois Han, un Papou et un européen – français), le groupe a effectivement obtenu un arbre phylogénétique qui fait des dénisoviens le groupe frère des néandertaliens et non plus une lignée antérieure à la divergence néandertal-sapiens figure 6).
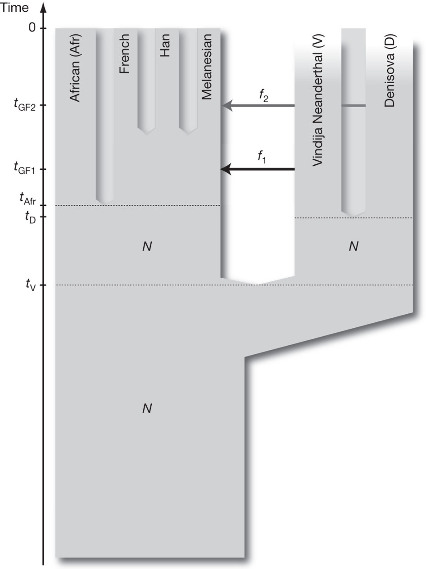
Cette comparaison entre génomes anciens et actuels montrait aussi que, comme cela avait déjà été démontré avec le génome néandertalien, des portions d'ADN dénisovien était présentes dans les séquences de certaines populations modernes : alors que des fragments d'ADN néandertalien apparaissent dans le génome de tous les humains non-africains, ceux d'ADN dénisovien se retrouvaient essentiellement dans les populations d'Asie, et plus encore dans le génome papou, c'est-à-dire chez les populations mélanésiennes d'Asie du Sud-Est. Les humains d'Asie possèdent donc à la fois des portions des génomes néandertalien et dénisovien. David Reich et ses collaborateurs proposèrent en conséquence un premier modèle d'histoire évolutive des populations paléolithiques eurasiennes incluant deux épisodes d'hybridation entre populations génétiquement distinctes : le première épisode d’hybridation entre les néandertaliens présents en Europe et en Asie occidentale et les sapiens qui y arrivaient à leur tour, sans doute depuis le Proche et le Moyen-Orient ; le second entre les dénisoviens qui occupaient probablement l'Asie orientale ou l'Asie du Sud-Est et des sapiens précédemment hybridés avec les néandertaliens et à l'origine, partiellement au moins, des actuelles populations mélanésiennes (figure 7). Ce schéma encore simple sera rapidement confirmé, complété, mais aussi complexifié par les recherches ultérieures.
Dans une dernière partie, le même article décrivait un autre fossile, cette fois une molaire (2e ou 3e ), dont les généticiens avaient réussi à tirer de l'ADN mitochondrial. Cette dent avait été déterrée dix ans plus tôt, en 2000, mais attribuée un peu trop vite à un Homo sapiens. Cet ADN s'avérait très peu différent (en 2 positions seulement) de celui extrait de la petite phalange, et nettement éloigné des ADNmt néandertalien et sapiens (380 différences recensées). Cette dent est donc dénisovienne, mais provient d'un autre individu que la phalange (ce dont attestait déjà sa taille et son état d'usure, qui permettait de l'attribuer à un ou une jeune adulte). Si les chercheurs n'en avaient pas encore tiré d'ADN nucléaire, cette dent devenait le premier fossile réellement informatif, au-delà de son contenu en ADN, sur les dénisoviens : sa morphologie, sa taille, l'épaisseur de son émail, le nombre et la forme de ses racines sont autant de paramètres par lesquels cette dent peut être comparée à celles d'autres hominines. Ses dimensions impressionnantes – avec une surface de la couronne de 13 mm sur 15 quand une molaire d'humain moderne ne dépasse guère 10 mm de large – suffisaient déjà à distinguer cette dent de celles des néandertaliens ou des humains modernes, et à la rapprocher plutôt des molaires d'hominines très anciens comme les Australopithèques ou Homo habilis.
Moins d'un an plus tard, en octobre 2012, Mathias Meyer et ses collaborateurs du Max Plank Institute dévoilaient un nouveau séquençage du génome dénisovien, considérablement plus précis que leur premier “brouillon” (avec cette fois une couverture de 30×), obtenu à l'aide d'une nouvelle méthodologie passant par la création d'une bibliothèque de fragments d'ADN simple brin au lieu d'ADN bicaténaire (Meyer et al., 2012[32]). Outre sa meilleure qualité, ce nouveau séquençage donnait des informations supplémentaires, notamment le niveau d'hétérozygotie de ce génome, c'est-à-dire la proportions d'allèles différents sur chaque paire de chromosomes. Cette hétérozygotie se révélait très faible, ce que les chercheurs ont interprété comme la conséquence génétique d'une population dénisovienne très réduite[5], une conclusion similaire à celle déduite des génomes néandertaliens (à l'hétérozygotie encore légèrement plus basse) : néandertaliens et dénisoviens ont manifestement eu des aires de répartition gigantesques (l'ensemble de l'Europe jusqu'à l'Altaï pour les premiers, tout le reste de l'Asie pour les seconds) mais des populations très réduites, autrement dit de très faibles densités de population (qui n'ont pourtant pas empêché les rencontres de ces formes humaines avec la nôtre lors des expansions d'Homo sapiens en Eurasie, peut-être à la faveur de l'exploitation des mêmes sites particulièrement intéressants, comme la grotte de Denisova). Cependant, l'ADN de Denisova 3 a aussi fait conclure aux généticiens que la répartition des dénisoviens pouvait être morcelée en petites populations, peut-être tout de même plus importantes (une centaine de personnes ou plus) que celles des néandertaliens, modélisées, elles, comme de tout petits groupes d'au plus soixante individus, émiettés et très isolés les uns des autres (Peyrégne et al., 2024[38]).
L'identification des dénisoviens et la découverte d'un deuxième épisode de métissage entre une forme humaine éteinte et une portion des ancêtres de l'humanité actuelle a évidemment suscité une floraison de nouvelles recherches, abordant les questions soulevées par cette humanité nouvelle par différentes approches.
Les paléontologues ont bien sûr cherché à identifier des fossiles qui pourraient représenter ces dénisoviens : en l'absence de tels fossiles caractéristiques, informatifs et suffisamment bien décrits pour servir de référence – c'est-à-dire définir l'holotype de l'espèce, cette forme humaine n'a pas pu recevoir de nom binominal linnéen dès sa découverte. D'autant plus que les fragments de fossiles à partir desquels elle avait été identifiée étaient non seulement trop petits ou trop peu informatifs, mais en plus irrémédiablement détruits dans l'extraction de leur matériel génétique.
Les généticiens (ou génomiciens) ont multiplié les analyses d'individus modernes pour mieux caractériser les portions de génome d'origine dénisovienne et modéliser la distribution de ces fragments d'ADN dans la population actuelle.
Les paléogénéticiens, quant à eux, ont continué leur recherche d'ADN ancien, mitochondrial et nucléaire, sur les restes fossiles disponibles. Les dénisoviens sont devenus un sujet parmi d'autres maintenant que les recherches en paléogénomique se sont multipliées, apportant des éclairages nouveaux sur des thématiques variées. Tout en s'efforçant d'optimiser les protocoles pour minimiser la quantité d'os, de dentine ou d'émail nécessaire à l'analyse, réduire encore les risques d'artefacts, identifier le mieux possible les fragments d'ADN ancien et exclure les contaminations, enfin tirer la plus grande quantité d'information possible des fragments récupérés, la paléogénétique a exploré les relations entre dénisoviens et sapiens, dénisoviens et néandertaliens, néandertaliens et sapiens, mais aussi entre sapiens. Ainsi, la comparaison des ADN anciens, retrouvés sur les restes archéologiques, avec ceux des humains d'aujourd'hui a confirmé l'existence de populations passées dont le génome n'a pas contribué à celui de l'humanité actuelle, autrement dit des branches d'Homo sapiens disparues sans descendance[6]. Les études menées sur des restes datés du Paléolithique supérieur et du Néolithique, puis des temps archéologiques plus récents comme l'Âge du bronze, ont profondément redessiné l'histoire des populations humaines, de leur différenciation et de leurs mélanges au fil des migrations (voir, par exemple, sur Planet-Vie, l’article La génomique humaine, une discipline qui se fait connaitre(lien externe - nouvelle fenêtre) de C. Langlois (2022[27]).
Pour en rester aux dénisoviens, la figure 8 montre l'évolution depuis 2010 du nombre de publications scientifiques retrouvées par le moteur de recherche de la plateforme Web of Science: Core Collection en entrant le mot-clé “denisovan*” dans la catégorie “sujet” (Topic) et en excluant les noms d'auteur commençant par “Denisov”. Même s'il n'est surement pas exhaustif, cet échantillon illustre l'expansion des études consacrées à cette forme d'humanité encore insoupçonnée il y a quinze ans.
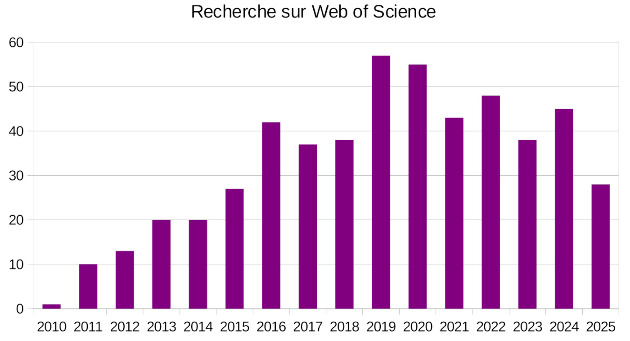
Source - © 2025 — Cyril Langlois
Recherche réalisée le 18 juillet 2025, les résultats fournis pour 2025 ne sont donc que partiels.
Denisova 11, l'hybride
Après 2010, les recherches menées dans la grotte de Denisova par les équipes du département de génétique évolutive de l'institut Max Planck de Leizig dirigé par Svante Pääbo n'ont pas cessé et ont régulièrement alimenté des publications dans les revues scientifiques les plus réputées, principalement Nature, faisant état de résultats aussi spectaculaires qu'inattendus. Ainsi en aout 2018, Nature dévoilait sur son site internet l'étude d'un nouveau fragment fossile, cette fois un os long (Slon et al., 2018[44]).
Première originalité, l'attribution de ce fragment d'os à un hominine plutôt qu'à un autre mammifère ne résultait pas d'une identification morpho-anatomique directe : l'os faisait partie d'une collection de plus de 2 000 restes trop fragmentaires pour être identifiés. C'est par l'analyse de son collagène (la principale protéine des tissus minéralisés), à l'aide d'une technique baptisée « empreintes de masse des peptides du collagène » (collagen peptides mass fingerprinting), relevant plus largement de la « zooarchéologie par spectrométrie de masse » (zooarcheology by mass spectrometry, ZooMS), qu'il a pu être attribué à une forme humaine. Les datations au radiocarbone l'estimaient âgé de plus de 50 000 ans (c'est-à-dire au-delà de la gamme d'âges accessible par la méthode du 14C). Au passage, grâce à la technique ZooMS, trois autres morceaux d'os ont pu être identifiés comme humains puis, par leur ADN, comme dénisoviens. Le nombre total de fossiles dénisoviens récoltés dans les deux chambres de la grotte de Dénisova a ainsi doublé, mais reste néanmoins très limité : six morceaux d'os, certains non-identifiables et désormais détruits lors des analyses, et trois dents.
De cette portion d'os long, les paléogénéticiens ont extrait et séquencé les génomes mitochondrial et nucléaire. L'individu, catalogué “Denisova 11”, a été ainsi identifié – par ses chromosomes sexuels – comme une jeune femme. Son génome mitochondrial s'apparentait à ceux des néandertaliens. Autrement dit, puisque l'ADN mitochondrial n'est transmis que par la lignée maternelle, de mère en fille, la mère de Denisova 11 était néandertalienne. Mais curieusement, cet ADNmt néandertalien se révélait proche de celui de néandertaliens plus récents, datés de 30 000 ans et découverts très loin de là, dans la grotte de Vindija, en Croatie. Il s'éloignait d'avantage de l'ADNmt des néandertaliens qui avaient pourtant occupé précédemment le site de Denisova ou d'autres abris voisins dans l'Altai.
Lorsque l'analyse s'est ensuite portée sur le génome nucléaire, celui-ci a montré un taux d'hétérozygotie étonnamment élevé. Seulement 40 % des fragments d'ADN se sont avérés apparentés aux séquences néandertaliennes ; près de 40 autres pour cents se rapprochaient, eux, du génome dénisovien. Cette dichotomie s'est confirmée quand les généticiens ont restreint leur comparaison aux sites du génome déjà identifiés comme distincts chez les néandertaliens et les dénisoviens. Denisova 11 s'est donc révélé être une hybride de première génération entre une mère néandertalienne et un père dénisovien, ce qu'aucun paléontologue ou paléogénéticien n'aurait jamais espéré découvrir, même si le laboratoire de l'institut Max Planck avait déjà identifié en 2015, parmi les fossiles de la grotte de Peștera cu Oase, en Roumanie, un individu sapiens dont l'un des aïeux, quatre à six générations plus tôt, était un néandertalien (Fu et al., 2015[14]).
La réunion, au même endroit – une cavité isolée au cœur de l'Asie sibérienne – de traces des trois populations humaines (dénisovien, néandertalien et sapiens) et d'un hybride interroge.
Elle suggère d'abord que cette grotte devait représenter une étape obligée, ou au moins un lieu de passage privilégié pour ceux qui pérégrinaient dans la région.
Elle pose aussi la question de la fréquence des hybridations : Denisova 11 était-elle un individu exceptionnel, identifiée par un incroyable coup de chance, ou un cas sinon fréquent, au moins récurrent ? L'examen détaillé des régions néandertaliennes du génome de Denisova 11 a révélé que certaines petites portions de ces séquences étaient homozygotes, c'est-à-dire que la mère néandertalienne et le père dénisovien portaient les mêmes variants génétiques à ces endroits-là. Dit autrement : le père dénisovien avait lui-même, parmi ses ancêtres lointains, des néandertaliens !
Dans le cas du métissage des sapiens avec les néandertaliens, les études ont souligné que si l'humain moderne a conservé quelques gènes néandertaliens, c'est parce qu'ils lui ont procuré alors un avantage sélectif ; mais la majorité des fragments génétiques néandertaliens semblent avoir été activement éliminés, donc au contraire contre-sélectionnés. Qu'en était-il chez les hybrides néandertaliens-denisoviens, deux groupes génétiquement plus proches l'un de l'autre que chacun d'entre eux avec Homo sapiens ?
Quoi qu'il en soit, il apparait qu'en dépit de populations réduites et dispersées et d'une divergence des lignées néandertalienne et dénisovienne entamée près de 360 000 ans plus tôt (vers −410 ka), l'hybridation est restée possible et féconde, comme elle l'était entre ces formes humaines d'Eurasie et les nouveaux venus sapiens. Ce qui, une fois encore, vient compliquer l'attribution taxonomique de ces humanités : sapiens et néandertal diffèrent par de nombreux caractères anatomiques et sans doute aussi cognitifs et culturels ; dénisovien et sapiens se distinguent génétiquement ; il parait délicat de rassembler ces groupes pour en faire des variétés d'une même espèce. Les désigner comme des espèces différentes parait légitime, même s'il faut renoncer au critère biologique d'Ernst Mayr, dont la pertinence a déjà été largement écornée par d'autres cas problématiques (ours brun et ours polaire, par exemple). Mais il semble que les études paléogénétiques progressent sans vraiment s'arrêter sur cette question de classification et de dénomination.
Les Dénisoviens, combien de divisions ?
Du côté de l'analyse génomique des populations actuelles, le séquençage d'un nombre croissant de génomes d'humain moderne a permis de mieux cerner la distribution géographique et la proportion de séquences génétiques dénisoviennes chez les populations d'Asie. Comme pour l'ADN néandertalien, le matériel génétique dénisovien ne représente qu'une très petite proportion du génome des humains d'aujourd'hui ; il est absent des populations autochtones du continent africain, de même que des humains d'Europe, dont les lointains ancêtres n'ont rencontré que les néandertaliens. On ne le trouve que chez les populations d'Asie, ainsi que chez les peuples qui ont atteint l'Amérique du Nord depuis la Sibérie en passant par le détroit de Béring lors de la dernière période glaciaire.
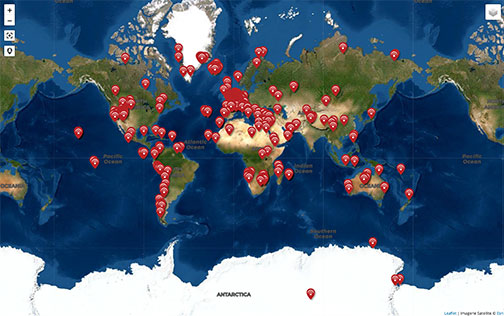
Mais curieusement, c'est parmi les peuples mélanésiens de Papouasie-Nouvelle Guinée que l'on rencontre l'une des plus forte fraction d'ADN dénisovien (presque 6 % du génome – figure 9). Les habitants des iles mélanésiennes, à l'Est de la « ligne de Wallace »[7] qui sépare deux provinces biogéographiques et passe entre Bornéo et Java à l'Ouest, les Philippines, Sulawesi (Célèbes) et Lombok à l'Est, présentent aussi un fort héritage néandertalien (2 %). La persistance d'ADN dénisovien dans cette partie du monde à l'extrémité occidentale de l'Asie a surpris. Ces portions d'ADN dénisovien s'avéraient aussi plus longs que ceux d'ADN néandertalien, ce qui signifie qu'ils n'ont pas été autant recombinés et fragmentés au fil des générations, donc que leur insertion dans le génome des sapiens (leur introgression) est plus récente. Si l'on suppose que les sapiens migrant vers la Mélanésie ont d'abord rencontré les néandertaliens, quelque part vers le Proche-Orient ou l'Europe de l'Est, puis les dénisoviens, ce résultat n'est pas étonnant. Mais l'écart de longueur moyenne entre les deux ADN archaïques (12 % de plus pour l'ADN dénisovien) plaide en faveur d'un écart temporel important entre les deux rencontres, autrement dit une hybridation avec les dénisoviens bien plus près de la Mélanésie, peut-être en Asie du Sud, il y a 44 000 à 59 000 ans (Reich, 2019[40]). La faible quantité d'ADN dénisovien chez les populations actuelles d'Asie et d'Asie du Sud-Est pourrait s'expliquer par les épisodes de migration et de déplacement survenus dans cette zone, au cours desquels des humains venant de l'Ouest, sans ADN dénisovien dans leur génome, se seraient mêlés aux autochtones ou les auraient remplacés, diluant l'héritage dénisovien. Des remaniements que n'auraient pas connu les populations néo-guinéennes, isolées au-delà de la ligne Wallace.
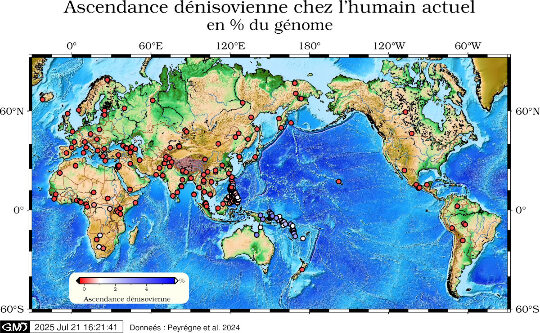
Source - © 2024 — Données Peyrégne et al. [ 38 ]
L'échelle de couleurs est choisie pour faire ressortir la situation particulière de la Mélanésie : la transition rouge-bleu correspond à la valeur de 1 %. Ces proportions d'ascendance archaïque sont estimées à l'aide de métriques statistiques spécifiques. Carte en projection de Mercator.
Cependant, ce scénario parait insuffisant. Il est contredit notamment par un fossile humain retrouvé non loin de Pékin, et daté de 40 000 ans, dont le génome ne montre pas de forte composante dénisovienne, comme on l'aurait attendu si l'hybridation sapiens-dénisovien s'était effectuée en Inde ou en Asie du Sud. Certains ont donc envisagé que le croisement des actuels autochtones néo-guinéens et des dénisoviens ait eu lieu en Asie du Sud-Est, voire sur les iles d'Indonésie ou de Malaisie. Mais jusqu'à présent, aucun fossile n'atteste de la présence d'hominine de grande taille dans cette région il y a 50 000 ou 60 000 ans (Reich, 2019[40]).
Pour autant, les populations d'Asie orientale et d'Asie du Sud portent encore, elles aussi, quelques traces d'ADN d'affinité dénisovienne (de l'ordre de 0,1 % ou moins). Et de même, dans les ADN récupérés sur des sapiens fossiles, “archaïques”, se retrouvent des séquences dont le degré de proximité avec les génomes dénisovien et néandertalien disponibles[8] implique des épisodes d'hybridation. Aujourd'hui, ces études conduisent à proposer qu'il existait plusieurs populations dénisoviennes, occupant différentes régions de l'Asie : les humains modernes se seraient hybridés avec l'une ou l'autre selon les endroits et les trajectoires de leur avancée vers l'Est. Une première étude publiée en 2018 avait identifié dans les génomes des populations actuelles d'Asie et d'Asie du Sud-Est deux types d'héritage dénisovien, l'un ressemblant plus que l'autre à la séquence de la phalange de Denisova. Les génomes des Chinois et des Japonais comprennent les deux, tandis que les Indiens, les Indonésiens ou les Papous ne présentent que le second : les groupes humains d'Asie orientale auraient donc connus deux introgressions dénisoviennes, ceux d'Asie du Sud et du Sud-Est une seule (Browning et al., 2018[4]). Les nouveaux séquençages réalisés depuis conduisent à des scénarios encore plus compliqués, envisageant au moins trois hybridations, avec deux épisodes séparés dans la lignée des actuels Papous (Ongaro et Huerta-Sanchez, 2024[35]). Enfin, le séquençage de la petite communauté Aetas des Philippines, des chasseurs-cueilleurs qui pourraient représenter les premiers sapiens arrivés dans cette région, a révélé une ascendance dénisovienne encore plus importante que celle des Papous, mais différente, autrement dit issue d'une quatrième population dénisovienne (Larena et al., 2021[28] ; Ongaro et Huerta-Sanchez, 2024[35]). Quant aux dates de ces multiples hybridations, elles sont évaluées sur la base du niveau de fragmentation de ces séquences dans les génomes actuels, mais restent imprécises et discutées.
L'héritage dénisovien chez Homo sapiens
Les séquences génétiques dénisoviennes insérées chez les humains modernes par ces croisements multiples ne représentent plus qu'une fraction infime du génome humain. Comme ceux des néandertaliens, les gènes dénisoviens semblent avoir été majoritairement expurgés du génome sapiens par la sélection naturelle. Ceux qui ont persisté ont dû, au contraire, se révéler utiles à la survie des humains dans les nouveaux environnements qu'ils découvraient en Asie. Les recherches se sont par conséquent penchées sur les fonctions des gènes d'origine dénisoviennes ou néandertaliennes. Dans les deux cas, le raisonnement de base est que les néandertaliens et les dénisoviens arpentaient les environnements d'Europe et d'Asie depuis des millénaires quand l'humain moderne y est arrivé à son tour. Par sélection naturelle, ces humanités cousines s'étaient adaptées à ces milieux. L'hybridation aurait alors permis aux Homo sapiens de s'adapter à leur tour, et plus rapidement, en récupérant les allèles favorables chez les populations autochtones (Singer, 2016[43]). Et, en effet, l'héritage néandertalien concerne entre autres des gènes impliqués dans la pigmentation de la peau ; les néandertaliens avaient une peau claire, absorbant suffisamment d'ultraviolets B pour synthétiser la vitamine D, alors que la fonction protectrice de la mélanine et d'une peau noire envers le rayonnement solaire ne leur était plus aussi nécessaire aux latitudes eurasiennes. Les populations d'Europe et d'Asie, confronté à des ensoleillements différents, n'ont d'ailleurs pas conservé les mêmes allèles néandertaliens associés à ces caractères cutanés (Sankararaman et al., 2016[42]). Les variantes génétiques néandertaliennes et dénisoviennes se retrouvent aussi dans les gènes des récepteurs Toll, impliqués dans la réponse immunitaire innée contre les bactéries, les champignons ou les parasites (Dannemann et al., 2016). Dès 2014, des travaux portant sur l'adaptation à l'altitude chez les Tibétains était arrivée à la conclusion qu'un gène, EPAS1, codant pour un facteur de transcription impliqué dans la réponse cellulaire à l'hypoxie et déjà repéré comme très fortement sélectionné dans ces peuplades, correspondait chez eux à un variant d'origine dénisovienne (Huerta-Sánchez et al., 2014[21]). Les Tibétains ne présentent pourtant que très peu d'ADN dénisovien. La conservation de ce variant particulier parait donc associée à un avantage sélectif. L'adaptation des Tibétains seraient ainsi passée par l'exploitation d'un variant génétique présent chez une ancienne humanité elle-même adaptée à de telles conditions. Mais en 2021, un nouvel article (Zhang et al., 2021[52]) a complexifié cette interprétation. Ses auteurs concluaient que :
- ce variant d'EPAS1 provenait d'une population dénisovienne « altaïque », proche de celle séquencée dans la grotte de Denisova, et dont on retrouve les traces chez les Asiatiques de l'Est, mais pas chez ceux du Sud ni chez les Néo-guinéens ;
- cette introgression serait ancienne, remontant à environ 48 000 ans ;
- mais la forte sélection du gène EPAS1 chez les Tibétains n'aurait eu lieu que beaucoup plus tard, il y a 8 900 ans seulement (peut-être quand les premiers chasseurs-cueilleurs tibétains ont commencé à occuper régulièrement ces régions inhospitalières). Autrement dit, pendant presque 40 millénaires, ce variant génétique serait resté sélectivement neutre et ne se serait révélé utile que bien après son insertion dans le génome des humains d'Asie. Mais dans ce cas, ce variant contribuait-il réellement déjà à l'adaptation à l'hypoxie chez les dénisoviens ? Et ces dénisoviens « altaïques » fréquentaient-ils effectivement déjà les hautes altitudes, à faible teneur en dioxygène, il y a 48 000 ans ? Avaient-ils développé cette adaptation, si c'en était déjà une, dans les montagnes de Sibérie et de Chine ou sur les hauteurs du Tibet ?
À côté de ce cas particulier très documenté, des allèles hérités des dénisoviens ont été détectés chez les Néo-Guinéens sur des gènes impliqués dans la réponse immunitaire, par exemple dans ceux du complexe majeur d'histocompatibilité (CMH), cet ensemble de gènes qui détermine les antigènes de surface des cellules et participent à la reconnaissance du soi et à la réponse immunitaire adaptative (Ongaro et Huerta-Sanchez, 2024[35]). Chez les Inuits d'Amérique du Nord, des allèles dénisoviens se retrouvent dans des gènes participant aux métabolisme des lipides et à la différenciation des adipocytes bruns et beiges, ces cellules graisseuses particulières productrices de chaleur (Ongaro et Huerta-Sanchez, 2024[35]). Là encore, on peut soupçonner une sélection positive de ces allèles chez ces nomades des milieux polaires. Et cela suggère à nouveau qu'au moins certaines populations de dénisoviens, celles de l'Altaï sibérien en l'occurrence, étaient adaptées aux environnements froids.
Reconstituer les dénisoviens sans (beau) fossile ?
Ainsi, la génétique et la paléogénétique ont tiré une moisson d'informations de l'exploitation du génome dénisovien découvert en 2010. Comme on l'a vu, leurs conclusions – qui ne restent, toutefois, que des interprétations basés sur des proximités statistiques entre séquences génétiques – concernent finalement d'abord l'évolution de notre propre espèce : les rencontres qu'elle a faite au cours de son expansion vers l'Est, les chemins qu'elle a pu suivre, les moyens par lesquels elle s'est adaptée à ces environnements, les répercussions que cet ADN étranger peut avoir sur la santé des individus d'aujourd'hui. En parallèle, et presque secondairement, elles renseignent sur les dénisoviens eux-mêmes : leur répartition géographique (immense), leur démographie (réduite et morcelée), quelques-unes de leurs adaptations. Mais elles nous disent peu, voire rien, sur ce à quoi ressemblaient ces individus. Jusqu'à récemment, ce que l'on savait de leur anatomie se résumait à la possession d'énormes molaires aux racines très écartées (à l'opposé de celle des néandertaliens). On pouvait éventuellement suspecter une certaine ressemblance avec les néandertaliens, puisque ces deux formes humaines partageaient un ancêtre commun direct. Cependant, dès l'obtention du génome dénisovien de référence (Meyer et al., 2012[32]), les paléogénéticiens avaient pointé la présence d'allèles qui, chez l'humain moderne, sont associés à une peau, des cheveux et des yeux sombres, alors que les néandertaliens, comme déjà dit, auraient plutôt eu la peau claire.
Y a-t-il moyen d'aller plus loin, en attendant de trouver des fossiles plus parlants ? Une équipe israélo-ibérique s'y est risquée en 2019, avec des résultats qui n'ont pas complètement convaincu la communauté scientifique. Leur approche méthodologique a consisté à examiner non pas les séquences génétiques proprement dites, mais la répartition dans le génome des modifications épigénétiques. Comme l'ont précisé ces chercheurs, les séquences génétiques ne suffisent pas : dénisoviens et humains modernes ne diffèrent que par moins de 100 substitutions non-synonymes dans les gènes codant pour des protéines et susceptibles de participer aux différences morpho-anatomiques (Gokhman et al., 2019[17]). Les 30 000 autres différences génétiques identifiées correspondent à des mutations synonymes ou non-codantes, donc sans effets phénotypiques. Certes, les techniques de séquençage génomique permettent désormais de chercher des corrélations entre certaines mutations ponctuelles (de type SNP) et des traits phénotypiques, par des études dites « d'association pangénomique » (genome-wide association studies, ou GWAS). Mais ces approches sont difficilement transposables aux dénisoviens. D'abord parce qu'elles nécessitent le génotypage de grandes cohortes d'individus. Ensuite parce qu'elles étudient la variabilité allélique liée à des mutations récentes au sein d'une même population (p. ex. les SNP associées à une teinte des cheveux chez les européens) et non les différences entre des groupes depuis longtemps séparés, comme dénisoviens et sapiens. Cette équipe a préféré reconstituer l'activité des gènes, partant du principe que celle-ci est régulée et modifiée par des processus épigénétiques, en particulier l'ajout de groupements méthyles (–CH3) sur certains nucléotides. Une méthylation importante dans la séquence d'un promoteur situé en amont d'un gène est généralement corrélée à une diminution d'activité ou à l'inhibition de ce gène.
Cependant, la méthylation elle-même n'est pas détectable sur l'ADN ancien. Les processus de dégradation de l'ADN la font disparaitre. Les chercheurs ont donc fondé leur cartographie sur les effets secondaires de cette modification épigénétique : les méthylations sont fréquentes dans les régions du génome où se suivent une cytosine et une guanine (succession dite « dinucléotide CpG » – le “p” indiquant la liaison phosphate). La cytosine y est alors méthylée en 5-méthylcytosine. Or, au cours des processus de dégradation post-mortem, la cytosine subit une désamination (perte d'un groupe amine –NH2) qui en fait un uracile, un nucléotide normalement absent de l'ADN, et que le séquenceur n'enregistre donc pas ; par contre, la 5-méthylcytosine désaminée devient une thymine, l'un des nucléotides habituels de l'ADN et le dinucléotide CpG devient TpG. Les régions de l'ADN ancien où la fréquence de ces TpG s'avère élevée sont donc probablement des zones qui, du vivant de l'organisme, étaient fortement méthylées, ce que l'équipe avait testé sur l'ADN néandertalien et sapiens dans un précédent article de 2014 (Gokhman et al., 2014[16]). Cette équipe a ainsi reconstitué la répartition des régions méthylées du génome dénisovien et l'a comparée à celle des humains actuels, des néandertaliens et des chimpanzés. Ils ont ensuite recherché dans les bases de données existantes les liens connus entre des caractéristiques phénotypiques et certains gènes, en supposant que la méthylation de ces gènes a des conséquences similaires aux mutations et modifie le phénotype.
David Gokhman et ses collègues ont concentré leurs examens sur les régions du génome différant d'au moins 50 % dans leurs niveaux de méthylation (Gokhman et al., 2019[17] ; Price, 2019[39]). Ils ont testé leur approche en vérifiant si les variations phénotypiques attendues d'après la répartition des méthylations dans l'ADN néandertalien et humain correspondaient effectivement aux différences constatées sur les squelettes de ces deux humanités. Les différences phénotypiques proposées par ces chercheurs restent toutefois seulement qualitatives et relatives : une face néandertalienne plus large que celle de l'humain moderne, un crâne plus bas, un pelvis plus large… Cependant, toutes les différences observées ne sont pas associées à des différences phénotypiques caractérisées et certaines variations supposées d'après les méthylations n'ont pas été confirmées pas les observations.
Appliqué à l'ADN dénisovien, ce protocole a abouti à une morphologie dénisovienne assez proche de celle des néandertaliens, avec un crâne bas, large et allongé, une mandibule très large, des dents à l'émail plus épais que le nôtre, des doigts plus longs aux extrémités plus larges. Ce dernier point est l'un des résultats qui ont mis en doute la validité de la méthode. Au début de ce même mois de septembre 2019, en effet, une équipe franco-russo-canadienne avait décrit la morphologie du doigt de la fillette dénisovienne Denisova 3. En analysant l'ADN mitochondrial d'une extrémité de doigt retrouvée dans la grotte de Denisova et en la reconstituant par ordinateur, elle avait démontré que ce fossile était le prolongement de la portion de phalange analysée en 2011 (Bennett et al., 2019[2]). Et, contrairement aux prédictions épigénétiques, la largeur de ce bout d'auriculaire le rapproche plus d'un doigt d'Homo sapiens que d'Homo neanderthalensis (figure 10). Sur ce critère et d'autres, la pertinence de la reconstitution proposée par l'analyse épigénétique a été mise en doute (Triou, 2019[45]). Sans oublier un biais majeur : les études génétiques comme épigénétiques restent fondées sur le génome d'une seule personne, qui plus est décédée jeune de cause inconnue… on fait mieux comme échantillon représentatif. Mais au-delà de cette controverse, l'intérêt de ce débat est d'illustrer comment les approches paléontologiques et moléculaires s'entrelacent désormais. Toute la puissance des analyses moléculaires n'est rien sans la mise au jour de fossiles, même très fragmentaires mais pourvoyeurs d'ADN ou de protéines ; et simultanément, le traitement des données moléculaires propose d'aller plus loin que la seule description morpho-anatomique des fossiles, moyennant des modèles et des hypothèses toujours discutables.
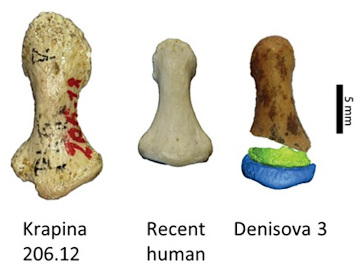
Source - © 2019 — Bennett et al. [ 2 ]
L'extrémité du doigt retrouvé est associé à une reconstitution virtuelle du fragment détruit dans l'extraction d'ADN qui a révélé l'existence des dénisoviens (en bleu et vert).
Image d’Eva-Maria Geigl, Institut Jacques Monod, France ; scan micro-tomographique et reconstitution virtuelle par Bence Viola, département d'anthropologie, Toronto, Canada.
Le retournement : des fossiles identifiés par leurs molécules
Alors que les paléogénéticiens développaient les outils moléculaires et informatiques qui leur ont permis d'extraire le maximum d'informations possibles de l'ADN du petit os Denisova 3, les paléontologues ont continué à chercher des fossiles susceptibles de représenter cette forme humaine inattendue. On l'a vu, des fragments d'os trouvés au même endroit ont pu être ajouté à la minuscule collection de spécimens dénisoviens. Mais les analyses génétiques suggèrent que l'Altaï sibérien n'était que l'une des régions d'Asie parcourues par les dénisoviens¨ ; La grotte de Dénisova n'était même peut-être que l'extrémité occidentale de leur domaine. D'après les reliquats dénisoviens détectés chez les humains actuels, on pouvait espérer trouver des fossiles dénisoviens de l'Altaï au Pacifique et de la Chine du Nord et la Sibérie jusqu'à la Mélanésie.
Les humains « archaïques » du Pléistocène supérieur d'Asie : un fouillis
Rapidement après l'identification moléculaire des dénisoviens, les paléontologues ont soupçonné que des fossiles déjà mis au jour en Asie et dont l'identification restait imprécise pouvaient peut-être correspondre à ces dénisoviens. D'autres fossiles ont également été trouvé depuis. Restait dans tous les cas à prouver cette affiliation dénisovienne.
Quelques fossiles, trouvés notamment en Chine et datés du Pléistocène moyen (étage Chibanien, de 774 000 à 129 000 ans avant le présent) et du Pléistocène supérieur (129 000 à 11 700 ans avant le présent), sont en effet imparfaitement identifiés, attribués à des taxons comme Homo heidelbergensis (un groupe devenu un fourre-tout pour les fossiles n'entrant ni dans la catégorie des H. erectus ni dans celle des H. neandertalhensis) ou Homo soloensis, ou à une catégorie encore plus floue d'« humains archaïques ». En 2017 s'y sont ajoutés deux crânes incomplets exhumés en 2007 et 2014 sur le site de Lingjing Xuchang (figure 12), près de la préfecture de Xuchang, dans la province chinoise du Henan et datés de 105 000 à 125 000 ans (Li et al., 2017[30]).
Chercher sans gène : l'alternative des protéines
Les dénisoviens n'étant défini que par leurs caractéristiques moléculaires, leur attribuer des fossiles exige de retrouver sur ces derniers les mêmes spécificités. En l'absence de caractéristiques morpho-anatomiques bien définies et consensuelles, hormis la taille et la forme des molaires, seules les données chimiques permettent d'identifier formellement un fossile dénisovien. Mais l'ADN est une molécule fragile, la remarquable conservation des molécules dans la grotte de Dénisova reste une exception. Néanmoins, en 2019, un fossile nettement plus substantiel qu'un bout de phalange et ne provenant pas de l'Altaï a pu être catalogué comme dénisovien. Il s'agit d'une demi-mandibule portant encore deux dents complètes et des racines dentaires (figure 11) (Chen et al., 2019[8]). Elle avait été trouvée en 1980 dans la grotte de Baishiya – située à 3 300 m d'altitude près de la localité de Xiahe, au Tibet – et conservée par des moines bouddhistes avant d'être récupérée par un géologue chinois. La datation de sa gangue carbonatée (par la méthode uranium-thorium) lui donne un âge de 160 000 ans.
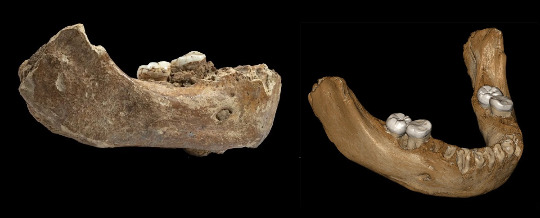
Les paléontologues chinois qui ont commencé à étudier ce fossile en 2016 ne sont pas parvenu à en extraire de l'ADN exploitable. Ils ont alors entamé une collaboration avec l'équipe du paléontologue Jean-Jacques Hublin de l'institut Max Planck de Leipzig. À défaut d'ADN, les chercheurs ont réussi à extraire de l'ivoire des dents des restes de protéines et à les analyser par spectrométrie de masse, par une méthode similaire à celle par laquelle a été isolé le morceau d'os de l'hybride Denisova 11. Les successions d'acides aminés de ces fragments de collagène ont pu être comparés à celles d'autres hominidés (humain, gorille, chimpanzé…), y compris les néandertaliens et les dénisoviens, en déduisant pour ces derniers leurs séquences protéiques (leur protéome) de leur séquences génétiques (leur génome). La mandibule “Xiahe 1”, s'est ainsi révélée clairement dénisovienne, ce que suggéraient déjà ses grandes molaires. L'équipe a également numérisé le fossile pour le nettoyer virtuellement et reconstituer la mandibule complète (figure 11), large, haute et sans menton. Cette mandibule prouve qu'en plein milieu de l'avant-dernière période glaciaire (il y a 190 000 à 130 000 ans), des dénisoviens occupaient les hautes altitudes tibétaines. Ce constat conduit évidemment à penser que c'est bien au Tibet que le variant dénisovien du gène EPAS1 a été sélectionné et, plus tard, récupéré et conservé par les humains modernes parvenus à leur tour sur ces hauteurs, une centaine de milliers d'années plus tard.
Enfin, des fouilles ultérieures dans la grotte de Baishiya y ont repéré, par la technique ZooMS, une côte humaine, que les données protéomiques ont à nouveau identifiée comme dénisovienne. Mais cet os est beaucoup plus récent que la mandibule, puisqu'âgé de seulement 48 000 à 32 000 ans (Xia et al., 2024[49]) ; les dénisoviens auraient donc fréquenté ces hauteurs durant des millénaires.
C'est encore cette méthode d'identification par les protéines plutôt que par l'ADN qui a permis d'étoffer le registre fossile dénisovien au cours des dernières années. Après celle de Xiahe en 2019, c'est une autre demi-mandibule qui a pu être déterminée ainsi. Là encore, l'histoire de ce fossile s'avère mouvementée ; il provient initialement des filets d'un bateau de pêche qui, en 2008 ou avant, raclait les profondeurs du détroit de Penghu, à 25 km de la côte occidentale de Taiwan (Tsutaya et al., 2025[46]). Les fonds de cette zone sont profonds de 60 à 120 m, ce qui signifie qu'ils étaient au moins en partie émergés au cours des dernières périodes glaciaires.
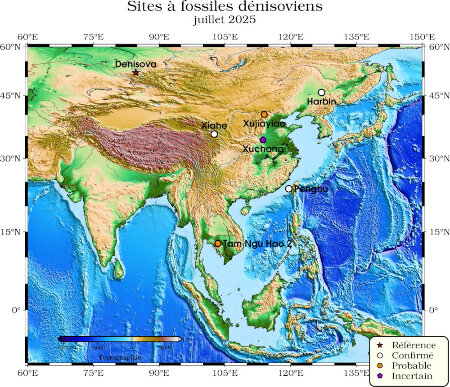
Source - © 2025 — Cyril Langlois
Du Nord au Sud : phalange et dents de la grotte de Denisova, en Russie, à 700 m d'altitude (Krause et al., 2010[25]) ; crâne de Harbin, Chine (Fu, Bai et al., 2025[12]) ; fossiles de Xujiayao, Cobra Cave, Chine, 1116 m (Xing et al., 2015[50]) ; mandibule de Xiahe, Baishiya Karst Cave, Tibet, 3280 m (Chen et al., 2019[8]) ; mandibule de Penghu, détroit de Taïwan (Tsutaya et al., 2025[46]) ; dents de Tam Ngu Hao 2, chaine des Annamites, Laos (Demeter et al., 2022[10]). Les fossiles de Xujiayiao et Tam Ngu Hao 2 sont rapprochés des dénisoviens par leurs analyses morphométriques. Les crânes de Xuchang pourraient également correspondre à des dénisoviens, mais cette attribution n'est pas encore démontrée (Li et al., 2017[30]).
Carte en projection cylindrique équivalente.
Le fossile fut acheté par un citoyen taiwanais, qui en fit don au Musée National de Science Naturelle de Taiwan. Un consortium de chercheurs taiwanais, japonais et australiens l'a finalement décrit en 2015 comme celle d'un « Homo archaïque » (Chang et al., 2015[7]), avant qu'une équipe internationale de chercheurs basés à Copenhague, Tokyo et Taiwan ne l'authentifie comme dénisovien en 2025.
La demi-mandibule, cataloguée “Penghu 1”, ressemble fortement à celle de Xiahe, dénichée 2 000 km plus à l'Est et 3 400 m plus haut, ce qui vient confirmer l'étendue de la distribution géographique des dénisoviens (figure 13). Comme celle de Xiahe également, elle porte encore des dents, qui constituent toujours des sources d'information précieuses. L'analyse protéomique a par ailleurs permis d'attribuer cette mâchoire à un individu masculin. Par contre, son âge reste mal contraint : son séjour sous-marin prolongé ne permet plus de le dater par les méthodes radio-chronologiques fondées sur l'uranium, aussi présent dans l'eau de mer. Les auteurs de la description de 2015 ont avancé deux intervalles plausibles, soit 190 000 à 130 000 ans, soit entre 70 000 et 10 000 ans.
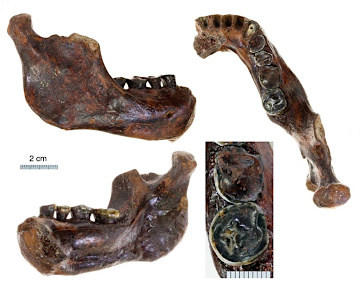
Source - © 2015 — Chang et al. [ 7 ]
Comme mentionné sur la figure 12, deux autres fossiles pourraient s'ajouter à cette collection de restes matériels dénisoviens : des dents encore insérées dans un morceau d'os maxillaire, provenant de la grotte de Xujiayao en Chine du Nord, entre 125 000 et 61 000 ans (Xing et al., 2015[50]) et une molaire provenant du Laos, sur le site de Tam Ngu Hao 2, 3 600 km plus au Sud, datée de 164 000 à 131 000 ans (Demeter et al., 2022[10]). L'examen morpho-anatomique de ces pièces, toutes deux datées du Pléistocène moyen, les rapproche de dents néandertaliennes, mais les en distingue aussi. Ce qui pourrait bien correspondre aux dents dénisoviennes.
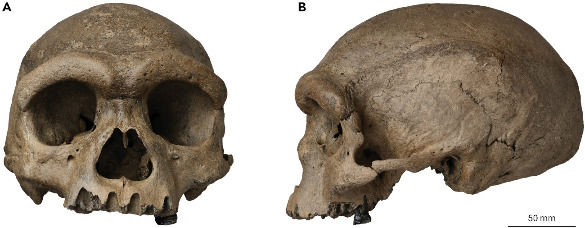
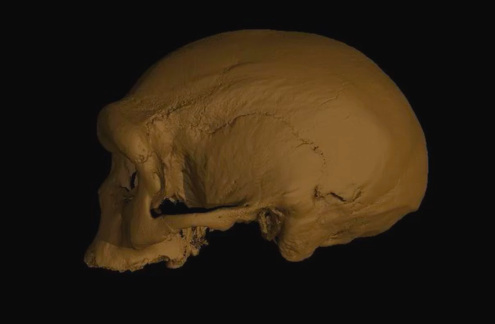
Mais le cas le plus spectaculaire, et qui vient donner une apparence concrète et expressive aux dénisoviens, est celui du crâne de Harbin, formellement identifié comme dénisovien en juin 2025. Ce crâne avait déjà défrayé la chronique paléo-anthropologique lors de sa présentation, en 2021, par une équipe de chercheurs chinois assistés de l'australien Rainer Grün et du paléoanthropologue Chris Singer du Muséum d'Histoire Naturelle de Londres (Ni et al., 2021[34]). Le fossile, très bien conservé, est un crâne de grand volume (1 420 cm3), comprenant la boite crânienne, la face et une molaire toujours insérée dans le maxillaire (figure 1). Cette découverte fut largement médiatisée alors (dans The Guardian, le New York Times, Le Monde…), d'abord pour le surnom d'« homme dragon » dont l'avait affublé les chercheurs, ensuite pour l'histoire qui l'accompagnait (Morin, 2021[33] ; Ni et al., 2021[34]). Selon celle-ci, ce crâne aurait été trouvé en 1933 par un ouvrier chinois travaillant à la construction d'un pont non loin de Harbin, 1 000 kilomètres au Nord-Est de Pékin, dans la Mandchourie alors envahie par le Japon (figure 12). Cet ouvrier aurait pris conscience de l'importance de sa trouvaille, peut-être en raison de la publicité qu'avait connu, quatre ans auparavant, la découverte du crâne de l'« homme de Pékin », un fossile aujourd'hui catalogué comme un Homo erectus. Il aurait alors caché sa découverte, en utilisant ce que les chercheurs ont décrit comme « une méthode chinoise traditionnelle de cacher les trésors », c'est-à-dire en l'enterrant dans un puits abandonné. Une fois les autorités chinoises rétablies dans la région, l'homme, redevenu fermier, aurait préféré taire sa participation aux travaux menés par l'occupant japonais et n'aurait ainsi jamais récupéré le fossile. Il n'en aurait dévoilé l'existence à ses descendants qu'à la veille de sa mort. L'un de ses petits-enfants aurait récupéré le crâne en 2018, puis aurait accepté de le confier à l'université de la province locale du Hebei. Toutefois, d'après la brève de la journaliste Dyani Lewis publiée dans Nature à l'occasion de la nouvelle publication de 2025, le chercheur Quian Ji auquel le fossile avait été confié soupçonne que la découverte soit due en réalité au petit-fils lui-même, qui ne l'aurait pas déclarée alors aux autorités locales (Lewis, 2025[29]).
Quoi qu'il en soit, le crâne de Harbin a d'abord été décrit en 2021 comme celui d'une nouvelle espèce d'hominine, baptisée Homo longi, et décrite comme plus proche d'Homo sapiens que d'Homo neanderthalensis. Sa datation radiochronologique par les séries de l'uranium lui donne un âge Pléistocène moyen de 146 000 ans. La stratigraphie de la région, quant à elle, le place dans une formation rocheuse datée de 309Ũ000 à 138 000 ans. Plusieurs chercheurs, dont le français Jean-Jacques Hublin, ont cependant presqu'aussitôt soupçonné que cet individu pouvait représenter un dénisovien. Il a été comparé à d'autres fossiles, notamment à la mandibule de Xiahe, mais aussi au crâne de Dali, découvert en 1978 dans la province du Shaanxi, à l'Ouest de Pékin (et jusque là attribué à H. heidelbergensis ou à H. erectus), ou encore à ceux de Xuchang. Des auteurs ont aussi pu proposer de diviser les fossiles chinois (Xiahe, Penghu, Xujiabao, Dali, Harbin, Xuchang…) en deux groupes distincts, les dénisoviens d'un côté – Dali… – et Harbin de l'autre (Bae et al., 2023[1]).
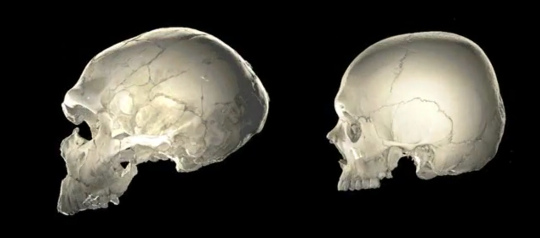
Les résultats publiés en juin 2025 viennent donc mettre un terme (provisoire) à ces discussions en attestant de l'appartenance du crâne de Harbin aux dénisoviens, et ce par deux méthodes indépendantes. D’une part, comme pour les mandibules de Xiahe et Penghu, par l'analyse protéomique, par spectrométrie de masse, de collagène extrait du crâne (Fu, Bai, et al., 2025[12]). D'autre part par le séquençage d'ADN mitochondrial extrait d'un calcul dentaire repéré sur la dent du crâne de Harbin – c'est-à-dire un morceau de plaque dentaire, ce biofilm bactérien minéralisé par le phosphate de calcium salivaire qui se développe à la surface des dents et demande un “détartrage” régulier quand notre brosse à dent ne suffit plus. Ce deuxième travail a donc réussi à récupérer dans ce calcul l'ADNmt de l'individu, à le séparer de l'ADN bactérien et des contaminations modernes, puis à le séquencer. L'analyse phylogénétique de cet ADNmt le place parmi les séquences dénisoviennes déjà connues (Fu, Cao, et al., 2025[13]). Le crâne de Harbin fournit donc un « visage » concret et un représentant fossile de grande qualité aux dénisoviens (figures 14 et 15), même s'il manque encore des éléments post-crâniens du squelette, susceptibles de confirmer ou d'infirmer les spéculations basées sur l'épigénétique. Les figure 16 et figure 17 suffisent à constater que ce crâne se distingue à la fois de celui d'un humain moderne et de celui d'un néandertalien, même s'il ressemble d'avantage à ce dernier, comme on pouvait s'y attendre d'après les résultats de la paléogénétique.
Avec cette confirmation se repose à nouveau la question de la dénomination taxonomique de ce groupe : le nom d'Homo longi attribué au crâne de Harbin en 2021 pourrait devenir le nom d'espèce des dénisoviens. Mais si, par exemple, le crâne de Dali ou d'autres fossiles s'avèrent également dénisoviens, par comparaison morphométriques ou moléculaires, des noms proposés antérieurement (Homo daliensis, Homo soloensis) pourraient le remplacer, en application de la règle d'antériorité. Mais la disparition du terme “dénisovien” au profit d'un nom d'espèce binominal sans lien avec le site de Dénisova pourrait susciter des réticences.
Quoi qu'il en soit, ces quinze années de recherche autour de ce groupe “fantôme” auront démontré la puissance des méthodes modernes de séquençage génétique et d'analyse protéomique. Elles ont abouti à une moissons d'informations sur cette population, sur la seule base des molécules exceptionnellement conservées dans quelques sites et quelques fragments, et dont la découverte a souvent résulté d'un heureux concours de circonstances. Ces années auront aussi vu l'établissement d'une nouvelle organisation de la recherche paléontologique des temps pléistocènes, fondée sur des coopérations internationales rassemblant paléontologues, paléogénéticiens et bio-informaticiens.
 Source - © 2021 — Chuang Zhao dans Ni et al. [ 34 ] |
 Source - © 2021 — Chuang Zhao dans Ni et al. [ 34 ] |
 Source - © 2018 — Philipp Gunz, Institut Max Planck Extrait de la brève d’Ewen Callaway, Nature News, 13 décembre 2018. |
 Source - © 2025 — Xijun Ni Extrait de la newsletter(lien externe - nouvelle fenêtre) Nature Briefing du 19 juin 2025. |
Comme illustré par la figure 14, les dénisoviens devaient produire des outils de pierre, à l'instar de leurs cousins néandertaliens et sapiens. Qu'en connait-on à l'heure actuelle ? La technique ZooMS appliquée aux débris osseux trouvés dans la grotte de Denisova a permis d'identifier des éléments dénisoviens et néandertaliens, et ainsi d'associer ces restes à quelques pierres taillées récoltées dans les couches archéologiques les plus profondes de la grotte. Ces outils lithiques pourraient donc être des réalisations dénisoviennes. Ce sont des grattoirs, des couteaux denticulés, des pointes, mais les chercheurs n'ont pas trouvé de biface comme pouvaient en produire les néandertaliens (Brown et al., 2022[3]). Hormis ces quelques éléments, les traits culturels des dénisoviens restent à découvrir.
Conclusions
Les dénisoviens constituaient une population d'hominines distincte mais directement apparentée aux néandertaliens. Leur divergence est datée entre 440 000 et 390 000 ans (figure 18).
Les dénisoviens ont été présents dans toute l'Asie au cours du Pléistocène moyen et récent, alors que les néandertaliens occupaient l'Europe et l'Asie occidentale jusqu'à l'Altai ; ces deux formes cousines devaient parcourir ces territoires en petits groupes, plus importants chez les dénisoviens que chez les néandertaliens, mais nettement isolés – génétiquement – les uns des autres.
Les hybrides néandertaliens-dénisoviens sont restées possibles, comme en témoigne l'ADN d'un os retrouvé sur le site de Denisova. Les hommes modernes se sont également hybridés avec les dénisoviens et en ont conservé des gènes avantageux, par exemple dans l'adaptation aux conditions hypoxiques des hautes altitudes tibétaines.
À l'aide des analyses protéomiques et génomiques, des fossiles plus conséquents que quelques dents et de petits os commencent à être attribués à cette humanité disparue. Après deux mandibules partielles, un crâne bien conservé, précédemment décrit comme une nouvelle morpho-espèce Homo longi, a été reconnu comme dénisovien en juin 2025.
L'aspect des dénisoviens reste énigmatique, même si le crâne de Harbin vient leur donner un visage. Des éléments de morpho-anatomie ont précédemment été proposés sur la base d'une cartographie des modifications épigénétiques du génome dénisovien, mais la validité de la méthode et ses conclusions sont discutées.
L'identification du crâne de Harbin ouvre la voie au ré-examen d'autres fossiles déjà répertoriés mais dont l'identification reste incertaine.
Les relations entre les néandertaliens, les dénisoviens et les taxons plus anciens, notamment Homo heidelbergensis, sont à ré-examiner, maintenant que l'on dispose d'un crâne dénisovien. D'autant que le statut d'Homo heidelbergensis fait débat depuis longtemps (Buck et Stringer, 2014). Si la divergence génétique entre néandertaliens et dénisoviens s'est effectuée il y a plus de 300 000 ans, Homo heidelbergensis peut-il encore être considéré comme l'ancêtre commun à ces deux populations alors que certains de ses fossiles sont plus récents ? Ou bien certains fossiles attribués à Homo heidelbergensis ne seraient-ils pas des dénisoviens ?
Enfin, les cultures matérielle et technologique des dénisoviens reste à peu près inconnue et se résume pour l'instant à quelques lames taillées.
Les outils de séquençage génétique et d'analyse protéomique disponibles aujourd'hui ouvrent la possibilité d'attester la présence des différentes formes d'hominines – ou d'autres organismes – sur un site même en l'absence de fossile reconnaissable, par l'analyse directe des sédiments, de fragments d'os, voire d'artefacts portés par les individus. Néanmoins, ces outils restent limités par la dégradation des acides nucléiques et des protéines, particulièrement dans les environnements chauds et humides des basses latitudes. Néanmoins, après une première transformation de la paléontologie induite par l'arrivée des outils géochimiques, notamment des analyses isotopiques, depuis une quarantaine d'années, une nouvelle évolution s'opère aujourd'hui avec l'intégration de la paléogénomique et de la paléoprotéomique. Le fossile réunit désormais autour de lui les sciences de la Terre, la physique, la chimie, la biologie évolutive… et la biologie moléculaire.
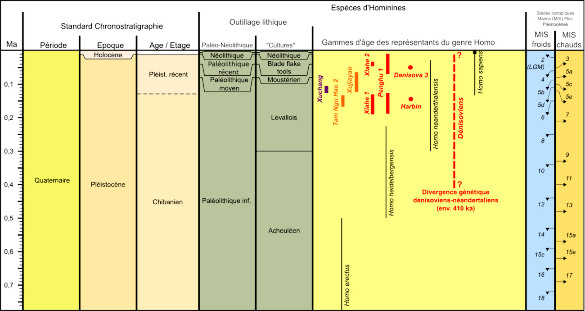
Source - © 2025 — Cyril Langlois
Charte chronostratigraphique réalisée avec l'application TimeScale Creator 8.1, modifiée et complétée sous Inkscape.
Bibliographie
- C.J. Bae, W. Liu, X. Wu, Y. Zhang, X. Ni, 2023. “Dragon man” prompts rethinking of Middle Pleistocene hominin systematics in Asia(lien externe - nouvelle fenêtre), The Innovation, 4, 6
- E.A. Bennett, I. Crevecoeur, B. Viola, A.P. Derevianko, M.V. Shunkov, T. Grange, B. Maureille, E.-M. Geigl, 2019. Morphology of the Denisovan phalanx closer to modern humans than to Neanderthals(lien externe - nouvelle fenêtre), Science Advances, 5, 9, eaaw3950 [Open access]
- S.Brown, D. Massilani, M.B. Kozlikin, M.V. Shunkov, A.P. Derevianko, A. Stoessel, B. Jope-Street, M. Meyer, J. Kelso, S. Pääbo, T. Higham, K. Douka, 2022. The earliest Denisovans and their cultural adaptation(lien externe - nouvelle fenêtre), Nature Ecology & Evolution, 6, 1, 28‑35
- S.R. Browning, B.L. Browning, Y. Zhou, S. Tucci, J.M. Akey, 2018. Analysis of Human Sequence Data Reveals Two Pulses of Archaic Denisovan Admixture(lien externe - nouvelle fenêtre), Cell, 173, 1, 53-61.e9 [Open archive]
- L.T. Buck, C.B. Stringer, 2014. Homo heidelbergensis(lien externe - nouvelle fenêtre), Current Biology, 24, , R214‑R215 [Open access]
- D. Caramelli, C. Lalueza-Fox, C. Vernesi, M. Lari, A. Casoli, F. Mallegni, B. Chiarelli, I. Dupanloup, J. Bertranpetit, G. Barbujani, G. Bertorelle, 2003. Evidence for a genetic discontinuity between Neandertals and 24,000-year-old anatomically modern Europeans(lien externe - nouvelle fenêtre), Proceedings of the National Academy of Sciences, 100, 11, 6593‑6597 [Free access]
- C.-H. Chang, Y. Kaifu, M. Takai, R.T. Kono, R. Grün, S. Matsu'ura, L. Kinsley, L.-K. Lin, 2015. The first archaic Homo from Taiwan(lien externe - nouvelle fenêtre) Nature Communications, 6, 6037
- F. Chen, F. Welker, C.-C. Shen, S.E. Bailey, I. Bergmann, S. Davis, H. Xia. H. Wang, R. Fischer, S.E. Freidline, T.-L. Yu, M.M. Skinner, S. Stelzer, G. Dong, Q. Fu, G. Dong, J. Wang, D. Zhang, J.-J. Hublin, 2019. A late Middle Pleistocene Denisovan mandible from the Tibetan Plateau(lien externe - nouvelle fenêtre), Nature, 569, 7756, 409‑412
- M. Dannemann A.M. Andrés, J. Kelso, 2016. Introgression of Neandertal- and Denisovan-like Haplotypes Contributes to Adaptive Variation in Human Toll-like Receptors(lien externe - nouvelle fenêtre), The American Journal of Human Genetics, 98, 1, 22‑33
- F. Demeter, C. Zanolli, K.E. Westaway, N. Joannes-Boyau, P. Duringer, M.W. Morley, F. Welker, P.L. Rüther, M.M. Skinner, H. McColl, C. Gaunitz, L. Vinner, T.E. Dunn, J.V. Olsen, M. Sikora, J.-L. Ponche, E. Suzzoni, S. Frangeul, Q. Boesch, P.-O. Antoine, L. Pan, S. Xing, J.-X. Zhao, R.M. Bailey, S. Boualaphane, P. Sichanthongtip, D. Sihanam, E. Patole-Edoumba, F. Aubaile, F. Crozier, N. Bourgon, A. Zachwieja, T. Luangkhoth, V. Souksavatdy, T. Sayavongkhamdy, E. Cappellini, A.-M. Bacon, J.-J. Hublin, E. Willerslev, L. Shackelford, 2022. A Middle Pleistocene Denisovan molar from the Annamite Chain of northern Laos(lien externe - nouvelle fenêtre), Nature Communications, 13, 1, 2557 [Open access]
- E. Essel, E.I. Zavala, E. Schulz-Kornas, M.B. Kozlikin, H. Fewlass, B. Vernot. M.V. Shunkov, A.P. Derevianko, K. Douka, I. Barnes, M.-C. Soulier, A. Schmidt, M. Szymanski, T. Tsanova, N. Sirakov, E. Endarova, S.P. McPherron, J.-J. Hublin, J. Kelso, S. Pääbo, M. Hajdinjak, M. Soressi, M. Meyer, 2023. Ancient human DNA recovered from a Palaeolithic pendant(lien externe - nouvelle fenêtre), Nature, 618, 7964, 328‑332 [Open access]
- Q. Fu, F. Bai, H. Rao, S. Chen, Y. Ji, A. Liu, E.A. Bennett, F. Liu F., Q. Ji, 2025a. The proteome of the late Middle Pleistocene Harbin individual(lien externe - nouvelle fenêtre), Science, 25. 389, 6761, 704-707
- Q. Fu, P. Cao, Q. Dai Q., E.A. Bennett, X. Feng, M.A. Yang, W. Ping, S. Pääbo, Q. Ji, 2025b. Denisovan mitochondrial DNA from dental calculus of the >146,000-year-old Harbin cranium(lien externe - nouvelle fenêtre), Cell, 188, 15, 3919-3926.e9 [Open access]
- Q. Fu, M. Hajdinjak, O.T. Moldovan, C. Constantin, S. Mallick, P. Skoglund N. Patterson, N. Rohland, I. Lazaridis, B. Nickel, B. Viola, K. Prüfer, M. Meyer, J. Kelso, D. Reich, S. Pääbo, 2015. An early modern human from Romania with a recent Neanderthal ancestor(lien externe - nouvelle fenêtre), Nature, 524, 7564, 216‑219
- G. Furelaud, 2004. Le séquençage des génomes(lien externe - nouvelle fenêtre), Planet-Vie
- D. Gokhman, E. Lavi, K. Prüfer, M.F. Fraga, J.A. Riancho, J. Kelso, S. Pääbo, E. Meshorer, L. Carmel, 2014. Reconstructing the DNA Methylation Maps of the Neandertal and the Denisovan(lien externe - nouvelle fenêtre), Science, 344, 6183, 523‑527
- D. Gokhman, N. Mishol, M. de Manuel, D. de Juan, J. Shuqrun, E. Meshorer, T. Marques-Bonet, Y. Rak, L. Carmel, 2019. Reconstructing Denisovan Anatomy Using DNA Methylation Maps(lien externe - nouvelle fenêtre), Cell, 179, 1, 180-192.e10 [Open archive]
- R. Higuchi, B. Bowman, M. Freiberger, O.A. Ryder, A.C. Wilson, 1984. DNA sequences from the quagga, an extinct member of the horse family(lien externe - nouvelle fenêtre), Nature, 312, 5991, 282‑284
- J.-J. Hublin, 2019. Denisova : notre cousin d'Asie sort de l'ombre, Pour la Science, 506, 28‑36
- J.-J. Hublin, 2021. Non, on ne vient pas de découvrir deux nouvelles espèces humaines, Pour la Science, 527, 54‑57
- E. Huerta-Sánchez, X. Jin X., Asan, Z. Bianba, B.M. Peter, N. Vinckenbosch, Y. Liang, X. Yi, M. He, M. Somel, P. Ni, B. Wang, X. Ou, Huasang, J. Luosang, Z.X.P. Cuo, K. Li, G. Gao, Y. Yin, W. Wang, X. Zhang, X. Xu, H. Yang, Y. Li, J. Wang, J. Wang, R. Nielsen, 2014. Altitude adaptation in Tibetans caused by introgression of Denisovan-like DNA(lien externe - nouvelle fenêtre), Nature, 512, 7513, 194‑197
- Z. Jacobs, B. Li, M.V. Shunkov, M.B. Kozlikin, N.S. Bolikhovskaya, A.K. Agadjanian, V.A. Uliyanov, S.K. Vasiliev, K. O'Gorman, A.P. Derevianko, R.G. Roberts, 2019. Timing of archaic hominin occupation of Denisova Cave in southern Siberia(lien externe - nouvelle fenêtre), Nature, 565, 7741, 594‑599
- Z. Jacobs, E.I. Zavala, B. Li, K. O'Gorman, M.V. Shunkov, M.B. Kozlikin, A.P. Derevianko, V.A. Uliyanov, P. Goldberg, A.K. Agadjanian, S.K. Vasiliev, F. Brink, S. Peyrégne, V. Slon, S. Pääbo, J. Kelso, M. Meyer, R.G. Roberts, 2025. Pleistocene chronology and history of hominins and fauna at Denisova Cave(lien externe - nouvelle fenêtre), Nature Communications, 16, 1, 4738 [Open access]
- Q. Ji, W. Wu, Y. Ji, Q. Li, X. Ni, 2021. Late Middle Pleistocene Harbin cranium represents a new Homo species(lien externe - nouvelle fenêtre), The Innovation, 2, 3, 100132 [Open access]
- J. Krause, Q. Fu, J.M. Good, B. Viola, M.V. Shunkov, A.P. Derevianko, S. Pääbo, 2010. The complete mitochondrial DNA genome of an unknown hominin from southern Siberia(lien externe - nouvelle fenêtre), Nature, 464, 7290, 894‑897 [Open access]
- J. Krause, L. Orlando, D. Serre, B. Viola, K. Prüfer, M.P. Richards, J.-J. Hublin, C. Hänni, A.P. Derevianko, S. Pääbo, 2007. Neanderthals in central Asia and Siberia(lien externe - nouvelle fenêtre), Nature, 449, 7164, 902‑904
- C. Langlois, 2022. La génomique humaine, une discipline qui se fait connaitre(lien externe - nouvelle fenêtre), Planet-Vie
- M. Larena, J. McKenna, F. Sanchez-Quinto, C. Bernhardsson, C. Ebeo, R. Reyes, O. Casel, J.-Y. Huang, K.P. Hagada, D. Guilay, J. Reyes, F.P. Allian, V. Mori, L.S. Azarcon, A. Manera, C. Terando, L. Jamero, G. Sireg, R. Manginsay-Tremedal, M.S. Labos, R.D. Vilar, A. Latiph, R.L. Saway, E. Marte, P. Magbanua, A. Morales, I. Java, R. Reveche, B. Barrios, E. Burton, J.C. Salon, M.J.T. Kels, A. Albano, R.B. Cruz-Angeles, E. Molanida, L. Granehäll, M. Vicente, H. Edlund, J.-H. Loo, J. Trejaut, S.Y.W. Ho, L. Reid, K. Lambeck, H. Malmström, C. Schlebusch, P. Endicott, M. Jakobsson, 2021. Philippine Ayta possess the highest level of Denisovan ancestry in the world(lien externe - nouvelle fenêtre), Current Biology, 31, 19, 4219-4230.e10 [Open access]
- D. Lewis, 2025. First ever skull from ‘Denisovan' reveals what ancient people looked like(lien externe - nouvelle fenêtre), Nature, 642, 8069, 848‑849
- Z.-Y. Li, X.-J. Wu, L.-P. Zhou, W. Liu, X. Gao, X.-M. Nian, E. Trinkaus, 2017. Late Pleistocene archaic human crania from Xuchang, China(lien externe - nouvelle fenêtre), Science, 355, 6328, 969‑972
- N. Longrich, 2025. Why one branch on the human family tree replaced all the others(lien externe - nouvelle fenêtre), Aeon Essays
- M. Meyer, M. Kircher, M.-T. Gansauge, H. Li, F. Racimo, S. Mallick, J.G. Schraiber, F. Jay, K. Prüfer, C. De Filippo, P.H. Sudmant, C. Alkan, Q. Fu, R. Do, N. Rohland, A. Tandon, M. Siebauer, R.E. Green, K. Bryc, A.W. Briggs, U. Stenzel, J. Dabney, J. Shendure, J. Kitzman, M.F. Hammer, M.V. Shunkov, A.P. Derevianko, N. Patterson, A.M. Andrés, E.E. Eichler, M. Slatkin, D. Reich, J. Kelso, S. Pääbo, 2012. A High-Coverage Genome Sequence from an Archaic Denisovan Individual(lien externe - nouvelle fenêtre), Science, 338, 6104, 222‑226
- H. Morin, 25 juin 2021. L'« homme dragon », un crâne chinois miraculeusement préservé(lien externe - nouvelle fenêtre), Le Monde
- X. Ni, Q. Ji, W. Wu, Q. Shao, Y. Ji, C. Zhang, L. Liang, J. Ge, Z. Guo, J. Li, Q. Li, R. Grün, C. Stringer, 2021. Massive cranium from Harbin in northeastern China establishes a new Middle Pleistocene human lineage(lien externe - nouvelle fenêtre), The Innovation, 2, 3, 100130 [Open access]
- L. Ongaro, E. Huerta-Sanchez, 2024. A history of multiple Denisovan introgression events in modern humans(lien externe - nouvelle fenêtre), Nature Genetics, 56, 12, 2612‑2622
- L. Orlando, R. Allaby, P. Skoglund, C. Der Sarkissian, P.W. Stockhammer, M.C. Ávila-Arcos, Q. Fu, J. Krause, E. Willerslev, A.C. Stone, C. Warinner, 2021. Ancient DNA analysis(lien externe - nouvelle fenêtre), Nature Reviews Methods Primers, 1, 14
- S. Pääbo, H. Poinar, D. Serre, V. Jaenicke-Després, J. Hebler, N. Rohland, M. Kuch, J. Krause, L. Vigilant, M. Hofreiter, 2004. Genetic Analyses from Ancient DNA(lien externe - nouvelle fenêtre), Annual Review of Genetics, 38, 645‑679 [Free]
- S. Peyrégne, V. Slon, J. Kelso, 2024. More than a decade of genetic research on the Denisovans(lien externe - nouvelle fenêtre), Nature Reviews Genetics, 25, 2, 83‑103
- M. Price, 2019. Face of the mysterious Denisovans emerges(lien externe - nouvelle fenêtre), Science, 365, 6459, 1232‑1232 [Full access]
- D. Reich, 2019. Comment nous sommes devenus ce que nous sommes - La nouvelle histoire de nos origines révélée par l'ADN ancien. 1re éd.(lien externe - nouvelle fenêtre), Quanto, EPFL PRESS, Lausanne, 384p. ISBN : 978-2-88915-308-4
- D. Reich, R.E. Green, M. Kircher, J. Krause, N. Patterson, E.Y. Durand, B. Viola, A.W. Briggs, U. Stenzel, P.L.F. Johnson, T. Maricic, J.M. Good, T. Marques-Bonet, C. Alkan, Q. Fu, S. Mallick, H. Li, M. Meyer, E.E. Eichler, M. Stoneking, M. Richards, S. Talamo, M.V. Shunkov, A.P. Derevianko, J.-J. Hublin, J. Kelso, M. Slatkin, S. Pääbo S., 2010. Genetic history of an archaic hominin group from Denisova Cave in Siberia(lien externe - nouvelle fenêtre), Nature, 468, 7327, 1053‑1060 [Open access]
- S. Sankararaman, S. Mallick, N. Patterson, D. Reich, 2016. The Combined Landscape of Denisovan and Neanderthal Ancestry in Present-Day Humans(lien externe - nouvelle fenêtre), Current Biology, 26, 9, 1241‑1247 [Open access]
- E. Singer, 2016. How Neanderthal DNA Helps Humanity(lien externe - nouvelle fenêtre), Quanta Magazine
- V. Slon, F. Mafessoni, B. Vernot, C. De Filippo, S. Grote, B. Viola, M. Hajdinjak, S. Peyrégne, S. Nagel, S. Brown, K. Douka, T. Higham, M.B. Kozlikin, M.V. Shunkov, A.P. Derevianko, J. Kelso, M. Meyer, K. Prüfer, S. Pääbo, 2018. The genome of the offspring of a Neanderthal mother and a Denisovan father(lien externe - nouvelle fenêtre), Nature, 561, 7721, 113‑116
- N. Triou, 2019. L'homme de Denisova, à un doigt d'Homo sapiens ?(lien externe - nouvelle fenêtre), Avec sciences, France Culture
- T. Tsutaya, R. Sawafuji, A.J. Taurozzi, Z. Fagernäs, I. Patramanis, G. Troché, M. Mackie, T. Gakuhari, H. Oota, C.-H. Tsai, J.V. Olsen, Y. Kaifu, C.-H. Chang, E. Cappellini, F. Welker, 2025. A male Denisovan mandible from Pleistocene Taiwan(lien externe - nouvelle fenêtre), Science, 388, 6743, 176‑180
- E. Van Dijk, C. Thermes, 2021. La révolution de la génomique : les nouvelles méthodes de séquençage et leurs applications(lien externe - nouvelle fenêtre), Planet-Vie
- E. Willerslev, A. Cooper, 2004. Review Paper. Ancient DNA(lien externe - nouvelle fenêtre), Proceedings of the Royal Society B: Biological Sciences, 272, 1558, 3‑16
- H. Xia, D. Zhang, J. Wang, Z. Fagernäs, T. Li, Y. Li., J. Yao, D. Lin, G. Troché, G.M. Smith, X. Chen, T. Cheng, X. Shen, Y. Han, J.V. Olsen, Z. Shen, Z. Pei, J.-J. Hublin, F. Chen, F. Welker, 2024. Middle and Late Pleistocene Denisovan subsistence at Baishiya Karst Cave(lien externe - nouvelle fenêtre), Nature, 632, 8023, 108‑113 [Open access]
- S. Xing, M. Martinón-Torres, J.M. Bermúdez de Castro, X. Wu, W. Liu, 2015. Hominin teeth from the early Late Pleistocene site of Xujiayao, Northern China(lien externe - nouvelle fenêtre), American Journal of Physical Anthropology, 156, 2, 224‑240
- E.I. Zavala, Z. Jacobs, B. Vernot, M.V. Shunkov, M.B. Kozlikin, A.P. Derevianko, E. Essel, C. De Fillipo, S. Nagel, J. Richter, F. Romagné, A. Schmidt, B. Li, K. O'Gorman, V. Slon, J. Kelso, S. Pääbo, R.G. Roberts, M. Meyer, 2021. Pleistocene sediment DNA reveals hominin and faunal turnovers at Denisova Cave(lien externe - nouvelle fenêtre), Nature, 595, 7867, 399‑403 [Open access]
- X. Zhang, K.E. Witt, M.M. Bañuelos, A. Ko, K. Yuan, S. Xu, R. Nielsen, E. Huerta-Sanchez, 2021. The history and evolution of the Denisovan-EPAS1 haplotype in Tibetans(lien externe - nouvelle fenêtre), Proceedings of the National Academy of Sciences, 118, 22, e2020803118 [Open access]
[1] Par exemple, la courte nouvelle de Maurice Leblanc L'homme à la peau de bique (1927) qui met en scène Arsène Lupin – hommage de Leblanc au Double meurtre dans la rue Morgue d'Edgar Alan Poe qui met, lui, en scène le détective Auguste Dupin ; et le feuilleton Balaoo (1911) de Gaston Leroux.
[2] On peut citer, parmi d'autres, le roman de SF de l'auteur américain Greg Bear, L'échelle de Darwin (1999) ; en français le thriller Régression de Fabrice Papillon (2019) ou la trilogie de SF Erectus de Xavier Muller (2018, 2021, 2022).
[3] L'estimation de la qualité d'un séquençage se fait, entre autres, par sa « couverture » C (coverage), qui compare le nombre N de séquences lues et la longueur moyenne L.de ces séquences à la taille totale du génome étudié G, on définit C = N × L / G. Une couverture de 2× pour un génome haploïde de 3000 bases signifie, par exemple, que le séquençage a trouvé quinze fois chaque position (N = 15) dans des segments d'une longueur moyenne de 400 nucléotides (L = 400), ou 10 fois dans des segments de 600 bases, etc., ce qui revient à dire que chaque base a été séquencée en moyenne deux fois.
[4] Toutes les comparaisons d'ADN évoquées ici se concentrent sur les différences ponctuelles des séquences nucléotidiques, autrement dit sur le niveau de polymorphisme des molécules d'ADN pour chaque nucléotide, ce que les généticiens qualifient de SNP (prononcé “snip”, Single Nucleotide Polymorphism), qui n'est que l'une des modifications possibles de la séquence génétique des chromosomes.
[5] En comparant le génome des humains actuels à ce nouveau génome dénisovien et à ceux des néandertaliens déjà disponibles, les généticiens de Leipzig ont aussi pu identifier des variants génétiques spécifiques à Homo sapiens, “fixés” dans la population sapiens (c'est-à-dire présents chez tous les humains) après la séparation entre Homo sapiens et l'ancêtre commun aux néandertaliens et aux dénisoviens.
[6] Comme ce fut probablement le cas des « sapiens archaïques » représentés par les fossiles les plus anciens attribués à notre espèce H. sapiens, mais qui n'ont pas toutes les caractéristiques anatomiques de l'humain moderne H. sapiens sapiens : l'Homme de Djebel Iroud, au Maroc, daté de 300 000 ans, celui d’Erto, en Éthiopie, de 196 000 ans, ceux de Qafzeh et de Skhul en Israël, datés de 140 000 et 90 000 ans, respectivement (Longrich, 2025[31]).
[7] Du nom d'Alfred Russell Wallace (1823-1913), qui l'a identifiée, voir l’article L'autre découvreur de la sélection naturelle : Alfred R. Wallace.
[8] À l'heure actuelle, les paléogénéticiens disposent de plusieurs génomes néandertaliens complets ; par contre, il n'y a toujours qu'un seul génome dénisovien de référence, celui de la phalange Denisova 3.